Capítulo 8 Bioenergética & Soluções Matriciais
- Estabilidade de biopolímeros;
- Interação ligante-receptor;
- Transporte de biomoléculas e íons;
- Mudanças conformacionais em biomacromoléculas;
- Associação de biopolímeros;
- Transferência de elétrons em proteínas;
- Combustão e síntese de biomoléculas;
- Geração de energia metabólica.
Para isto é necessário determinar-se o valor de \(\Delta\)G da transição de fase, o que pode ser realizado de diversas maneiras, e a partir de metodologia igualmente diversificada. Assim, por meio de medidas espectroscópicas (absorção molecular, fluorescência, luminescência), hidrodinâmicas (viscosimetria, coeficiente de sedimentação, pressão osmótica), eletroquímicas (potenciometria, voltametria), ou de atividade biológica, dentre muitas, é possível quantificar o parâmetro termodinâmico \(\Delta\)G. Esse, por sua vez, pode ser extraído das relações que seguem, considerando-se uma transição de dois estados:
Onde \(K_{eq}\), [D], e [N] representam, respectivamente, a constante de equilíbrio de desnaturação da proteina, bem como as concentrações dessa na forma desnaturada e nativa.
Em \(35^{o}\)C: \(\Delta\)G\(_{desn}\) = \(\Delta\)H\(_{desn}\) - 308 * \(\Delta\)S\(_{desn}\) = +4,4 kJ \(mol^{-1}\)
Em \(45^{o}\)C: \(\Delta\)G\(_{desn}\) = \(\Delta\)H\(_{desn}\) - 318 * \(\Delta\)S\(_{desn}\) = -5,2 kJ \(mol^{-1}\)
Outra solução, mais prática e computacional, envolve a resolução do sistema de equações lineares, tal como segue:
Onde \(x_1\) e \(x_2\) representam, respectivamente, \(\Delta\)H\(_{desn}\) e \(\Delta\)S\(_{desn}\).
\[A = \begin{bmatrix} a_{11} & a_{12}\\ a_{21} & a_{22} \end{bmatrix} , \]
\[x = \begin{bmatrix} b_1\\ b_2 \end{bmatrix} , \]
\[b = \begin{bmatrix} x_1\\ x_2 \end{bmatrix} \]
8.1 Solução de sistema de equações lineares no R
R, define-se inicialmente a matriz para A e a matriz para b tal que:\[A = \begin{bmatrix} 1 & -308\\ 1 & -318 \end{bmatrix} , \]
\[b = \begin{bmatrix} +4,4\\ -5,2 \end{bmatrix} \]
# Definição de matrizes
A = matrix(c(1,-308,1,-318),ncol=2,byrow=TRUE) # matriz A; o sinal negativo decorre da função possuir inclinação negativa
b = matrix(c(4.4,-5.2),ncol=1) # matriz bsolve:# Solução matricial para sistema linear
solve(A) %*% b # # a operação %*% indica o produto escalar de dois vetores ("dot product")## [,1]
## [1,] 300
## [2,] 1\[\begin{equation} \begin{pmatrix} 1 \\ 2 \\ 3 \\ \end{pmatrix} * \begin{pmatrix} 1 & 2 & 3 \\ \end{pmatrix} = \begin{pmatrix} 1 & 2 & 3 \\ 2 & 4 & 6 \\ 3 & 6 & 9 \\ \end{pmatrix} \tag{8.9} \end{equation}\]
Dessa forma, tal como visto no capítulo Enzimas, poderíamos solucionar os parâmetros \(\Delta\)H\(_{desn}\) e \(\Delta\)SH\(_{desn}\) por ajuste linear. De fato, uma das expressões de Van’t Hoff que definem esta relação linear é:
De fato, a solução matricial de ajuste linear pode ser obtida a partir da relação abaixo:
lm sejam extraídos por outras funções do algoritmo de cálculos matriciais/estatísticos. Na equação (8.11) o termo entre parênteses envolve a operação de inversão da matriz. Em álgebra linear não existe a operação de divisão para matrizes, mas somente a multiplicação de uma matriz por um escalar ou pela inversa de outra. E mesmo assim, somente se tratar-se de uma matriz quadrada. Dessa forma o termo (X\(^{T}\) X)\(^{-1}\) só pode ser calculado com inversão matricial. No R essa ação é dada pelo comando solve.Com dantes, é vital importância também que a matriz X contendo a variável independente seja criada com valores unitários à esquerda, tal como segue:
\[X = \begin{bmatrix} 1 & x_{1}\\ 1 & x_{2}\\ 1 & x_{3}\\ ... & ... \end{bmatrix} \]
# Repetindo os dados para as variáveis de Lineweaver-Burk
S=seq(0.1,1,length.out=20) # gera uma sequência com 20 pontos entre 0 e 1 para valores de substrato
Vm=10; Km=0.5 # parâmetros cinéticos
set.seed(1500) # estabelece a mesma semente aleatória do gráfico direto de Michaelis-Menten, para reproducibilidade dos pontos
erro=runif(20,0,1) # comando para erro uniforme (no. de pontos, min, max)
v=Vm*S/(Km+S)+erro # equação de Michaelis-Menten
inv.S=1/S # cria variáveis para o duplo-recíproco
inv.v=1/v
# Criação das matrizes A e b
A2=matrix(c(rep(1,20),inv.S),nrow=20,byrow = FALSE) # cria matriz com valor unitário necessário antes da variável independente
b2=as.matrix(inv.v,nrow=1,byrow=FALSE) # vetor b
# Solução matricial do ajuste linear
beta = solve(t(A2) %*% A2) %*% t(A2) %*% b2; beta## [,1]
## [1,] 0.11
## [2,] 0.03lm do R.8.2 Matrizes e R
Dessa forma, é interessante uma rápida panorâmica no potencial de matrizes que o
R possui.# Algumas manipulações com matrizes
## Identificação de linhas e colunas
res = matrix(c(-308,-318),nrow=2,byrow=TRUE, # definição de matriz
dimnames = list(c("Delta H","Delta S"),"kJ/mol"))
res## kJ/mol
## Delta H -308
## Delta S -318## Operações aritméticas
m1 = matrix(c(1,2,3,4), nrow=2, byrow=T)
m2 = matrix(c(4,5,6,7),nrow=2, byrow=T)
m1 + 5; m2-7 # soma ou subtração em escalar## [,1] [,2]
## [1,] 6 7
## [2,] 8 9## [,1] [,2]
## [1,] -3 -2
## [2,] -1 0m1^2; sin(m2) # potência ou trigonometria## [,1] [,2]
## [1,] 1 4
## [2,] 9 16## [,1] [,2]
## [1,] -0.8 -1.0
## [2,] -0.3 0.7m1 + m2 # soma de elementos em matrizes de igual dimensão## [,1] [,2]
## [1,] 5 7
## [2,] 9 11m1 * m2 # multiplicação de elementos em matrizes de igual dimensão## [,1] [,2]
## [1,] 4 10
## [2,] 18 28m1 %*% m2 # produto cruzado de matrizes ("dot product" de vetores)## [,1] [,2]
## [1,] 16 19
## [2,] 36 43det(m1) # determinante da matriz## [1] -2t(m2) # transposição da matriz## [,1] [,2]
## [1,] 4 6
## [2,] 5 7diag(m1) # matriz diagonal## [1] 1 4solve(m2) # inverso da matriz## [,1] [,2]
## [1,] -3 2
## [2,] 3 -2eigen(m1) # autovalor ("eingenvalue") e autovetor ("eigenvector") da matriz## eigen() decomposition
## $values
## [1] 5.4 -0.4
##
## $vectors
## [,1] [,2]
## [1,] -0.4 -0.8
## [2,] -0.9 0.6R também permite várias outras operações utilizadas em cálculo numérico e simbólico que utilizam matrizes, tais como as funções kronecker (multiplicação matricial), svd (Single Decomposition Value), qr (Decomposição QR), e chol(Decomposição de Cholesky).8.3 Solução de parâmetros termodinâmicos de estabilidade enzimática
O formalismo dessa análise passa pela Teoria das Colisões de Arrhenius, bem como pela Teoria do Estado de Transição de Eyring, resultando no sistema linear de equações como o que segue:
Onde os termos com \(\ddagger\) simbolizam as variações de quantidades referentes à ativação (ou desativação) da enzima (estado de transição do complexo ativado), \(K_{B}\) representa a constante de Boltzmann (1,381x10\(^{-23}\) JK\(^{-1}\)), h a constante de Planck (6,686x10\(^{-34}\) J*s), e R a constante geral dos gases (8,314 JK\(^{-1}\) mol\(^{-1}\)). Nem sempre é possível convergir uma solução matricial pela simples utilização de multiplicação cruzada (dot product). | O trecho de código abaixo exemplifica essa tentativa, a partir dos dados publicados por Riaz e cols (Bhatti et al. 2007) abaixo, e considerando previamente as matrizes A e b em função dos parâmetros explicitados pelos autores:
\[\begin{equation} \Delta G^{\ddagger}=65920\, J\,mol^{-1} \\ T = 328 K \\ kcat = 217 s^{-1} \tag{8.13} \end{equation}\]\[A = \begin{bmatrix} 1 & -328\\ -3.67e-4 & 0.120\\ \end{bmatrix} , \]
\[b = \begin{bmatrix} 65920\\ -24.17\\ \end{bmatrix} \]
# Tentativa de solução matricial simples em dados publicados (Appl. Microbiol. Biotechnol, 73, 1290, 2007):
A = matrix(c(1, -3.67e-4,-328,0.120),nrow=2, byrow=TRUE)
b = matrix(c(65921,-24.17),nrow=2)
solve(A) %*% b## [,1]
## [1,] -2e+07
## [2,] -6e+10solve também sofre de resolução para o problema, incorrendo em erro se executada, tal como no trecho que segue. Observe também a possibilidade distinta para a construção das matrizes.Aframe <-data.frame(c(1,-3.67e-4),c(-328,0.120))
A <- as.matrix(Aframe)
b <- as.matrix(c(65921,-24.17))
solve(t(A)%*%A)%*%t(A)%*%bR, tal como o rootSolve já utilizado ou o nleqslv empregado anteriormente. Nesse sentido, a minimização de busca de raízes para o sistema de equações pode ser demonstrada a seguir:library(rootSolve)
T = 328
R = 8.314
h = 6.626e-34
kb = 1.381e-23
kcat = 217
DG = -R*T*log((kcat*h)/(kb*T)) # 65920 J/mol
model=function(x) c(x[1]-T*x[2]-DG, x[2]/R-x[1]/(R*T)-log((kcat*h)/(kb*T)))
(ss=multiroot(model,start=c(50000,50000)))## $root
## [1] 40844 -76
##
## $f.root
## [1] 2e-09 -8e-13
##
## $iter
## [1] 3
##
## $estim.precis
## [1] 1e-09Comparando-se os valores, os autores encontraram \(\Delta\) H\(^{\ddagger}\) = 33,3 kJmol\(^{-1}\) e \(\Delta\) S\(^{\ddagger}\) = -99,8 Jmol\(^{-1}\)K\(^{-1}\). Perceba a semelhança dos resultados obtidos pela minimização de raízes com os parâmetros encontrados pelos autores. A obtenção do valor de \(\Delta\) H\(^{\ddagger}\) para esses, contudo, foi obtida somente a partir da obtenção do valor experimental de energia de ativação de Arrhenius (Ea), pela inclinação de um gráfico linearizado da taxa de reação, tal como segue:
require(rootSolve)
T = 328
R = 8.314
h = 6.626e-34
kb = 1.381e-23
kcat = 217
DG = 66000
model=function(x) c(x[1]-T*x[2]-DG, x[2]/R-x[1]/(R*T)-log((kcat*h)/(kb*T)))
(ss=multiroot(model,start=c(50000,50000)))## $root
## [1] 5e+10 2e+08
##
## $f.root
## [1] 177.51 -0.09
##
## $iter
## [1] 3
##
## $estim.precis
## [1] 898.4 Entalpia De Reação Por Matrizes
Matematicamente a Lei de Hess pode ser expressa como:
Onde \(\nu\) representa a estequiometria da reação, ou seja, o número de mols de cada composto/elemento, enquanto P e R referem-se à Produto e Reagente.
Para isso, é necessário 1) compor as matrizes A e b, 2) calcular o vetor de coeficients beta, e 3) efetuar o produto escalar (%*%) de beta com uma matriz formada pelos valores de entalpia de formação. O racional para compor as matrizes envolve elencar cada composto com sua estequiometria de reação, e exige que para reagentes seja conferido valor negativo, enquanto que para produtos, valor positivo.
A tabela abaixo ilustra essa construção para o problema em questão.
library(knitr) # para gerar a tabela
comp<-c("C2H2","O2","CO2","H2O","C2H6","H2") # elenco dos compostos envolvidos
rea1<-c(-2,-5,+4,+2,0,0) # estequiometria (reação1)
rea2<-c(0,-7,+4,+6,-2,0) # estequiometria (reação2)
rea3<-c(0, -0.5, 0,+1,0,-1) # estequiometria (reação3)
incog<-c(-1,0,0,0,+1,-2) # estequiometria da reação com entalpia desconhecida
tab_esteq<-data.frame(comp,rea1,rea2,rea3,incog) # dataframe com os resultados
colnames(tab_esteq)<-c("composto","reação 1","reação 2", "reação 3", "etano") # nomeia as colunas
knitr::kable(tab_esteq, caption="Estequiometria reacional para solução matricial de formação de etano (C2H6).", "pipe") # tabela| composto | reação 1 | reação 2 | reação 3 | etano |
|---|---|---|---|---|
| C2H2 | -2 | 0 | 0.0 | -1 |
| O2 | -5 | -7 | -0.5 | 0 |
| CO2 | 4 | 4 | 0.0 | 0 |
| H2O | 2 | 6 | 1.0 | 0 |
| C2H6 | 0 | -2 | 0.0 | 1 |
| H2 | 0 | 0 | -1.0 | -2 |
# Solução matricial para entalpia de formação
A=matrix(c(-2,0,0,-5,-7,-0.5,4,4,0,2,6,1,0,-2,0,0,0,-1), nrow=6, byrow=T) # cria matrix das reações com variação de entalpia conhecida
b=matrix(c(-1,0,0,0,1,-2),nrow=6,byrow=T) # cria matriz dos coeficientes estequiométricos da reação com variação de entalpia desconhecida
beta = solve(t(A) %*% A) %*% t(A) %*% b # cálcula beta
energ=matrix(c(-2600,-3210,-286),nrow=1,byrow=T) # cria matriz com os valores de entalpia
etano=energ %*% beta
cat("Valor para deltaHr etano: ", etano, " kJ/mol")## Valor para deltaHr etano: -267 kJ/mol8.5 Quantidades Termodinâmicas Por Ajuste Polinomial
Não obstante, não poderíamos utilizar as relações lineares de matrizes ou ajustes lineares para solucionar parâmetros quantitativos em situações que não repousassem no comportamento linear entre as variáveis, tal como referido pela equação (8.10), por exemplo.
T=c(5.29,10.07,15.23,20.21,25.11,30.29,37.39)+273 # dados de temperatura, em graus Kelvin
dG=c(11.74,12.17,12.46,12.73,12.88,12.98,13.13)*-1e3 # dados de -deltaG, em kcal/mol
plot(T,dG,
xlab = "T, K", ylab=expression(paste(Delta,"G, kcal/mol"))
)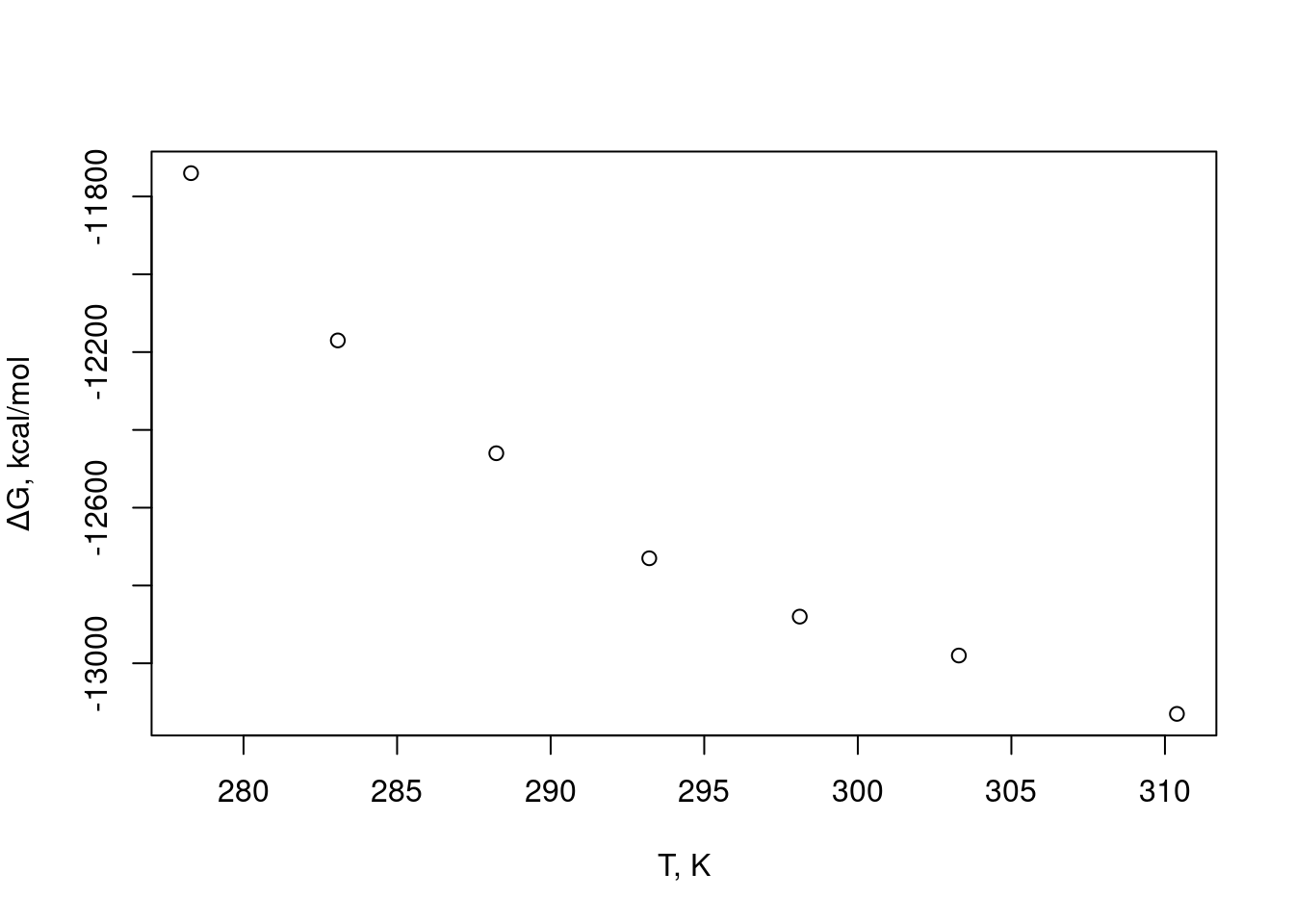
Figura 8.1: Dependência da temperatura com a variação de energia de Gibbs da interação de insulina com seu receptor.
pol3 <- lm(dG~poly(T,3,raw=TRUE)) # ajuste a polinômio de 3o. grau; "raw=TRUE" é essencial
# Alternativamente, pode-se também ajustar polinômios como
# pol3<-lm(dG ~ T + I(T^2)+I(T^3))
summary(pol3)##
## Call:
## lm(formula = dG ~ poly(T, 3, raw = TRUE))
##
## Residuals:
## 1 2 3 4 5 6 7
## 2.35 -13.43 26.73 -16.41 -8.58 12.16 -2.83
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 8.97e+05 2.45e+05 3.66 0.035 *
## poly(T, 3, raw = TRUE)1 -8.84e+03 2.50e+03 -3.54 0.038 *
## poly(T, 3, raw = TRUE)2 2.87e+01 8.49e+00 3.37 0.043 *
## poly(T, 3, raw = TRUE)3 -3.11e-02 9.61e-03 -3.23 0.048 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 22 on 3 degrees of freedom
## Multiple R-squared: 0.999, Adjusted R-squared: 0.998
## F-statistic: 1.04e+03 on 3 and 3 DF, p-value: 5.02e-05plot(T,dG,
xlab = "T, K", ylab=expression(paste(Delta,"G, kcal/mol"))
# gráfico de T x deltaG
)
lines(T,fitted(pol3),col="red") # curva ajustada sobre os dados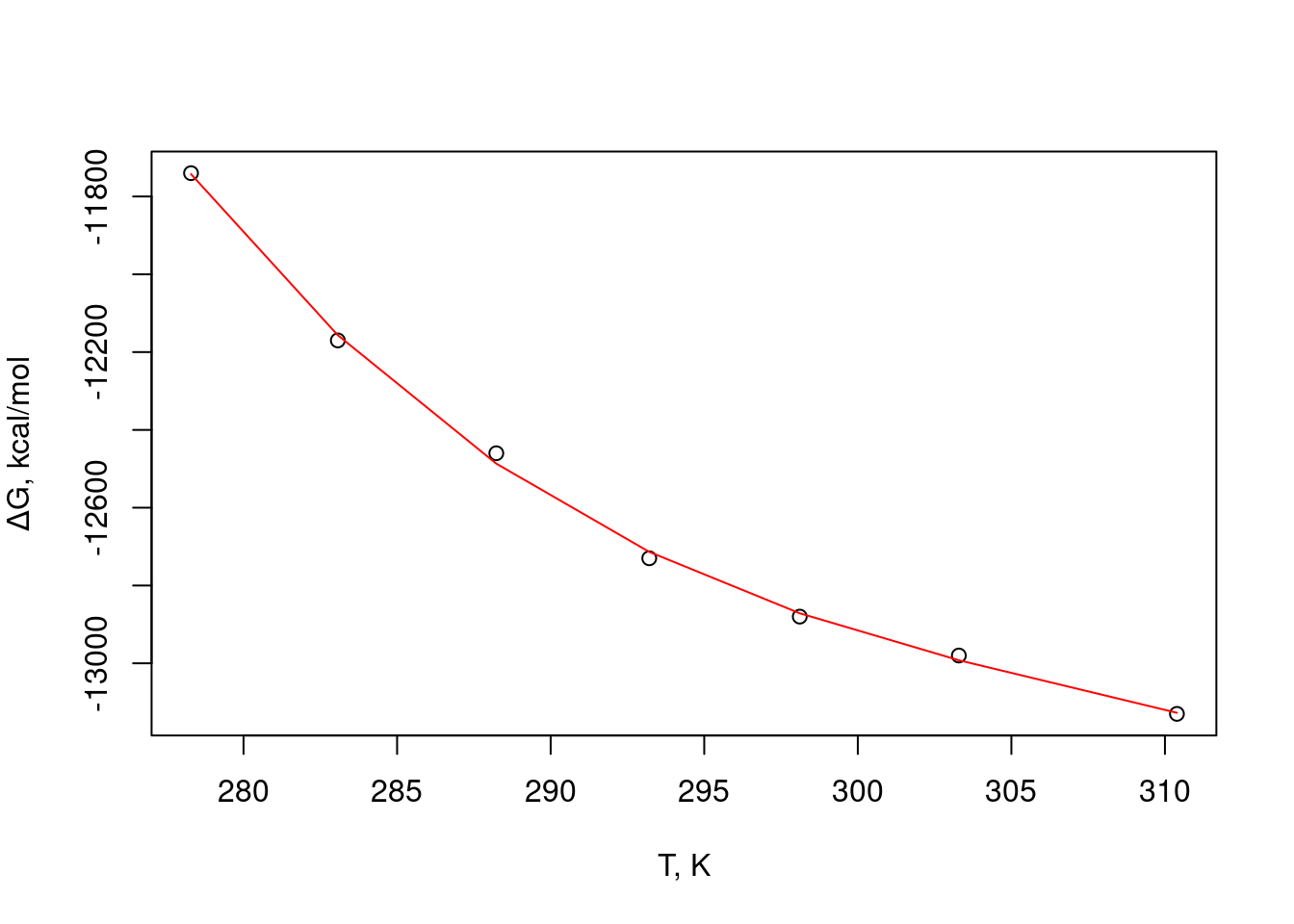
Figura 8.2: Ajuste polinomial sobre os dados de variação de energia de Gibbs da interação de insulina com seu receptor.
\[A = \begin{bmatrix} 1 & 278.29\\ 1 & 283.07\\ 1 & 288.23\\ 1 & 293.21\\ 1 & 298.11\\ 1 & 303.29\\ 1 & 310.39\\ \end{bmatrix} , \] \[b = \begin{bmatrix} -11740\\ -12170\\ -12460\\ -12730\\ -12880\\ -12980\\ -13130\\ \end{bmatrix} \]
\[matriz \,V = \begin{bmatrix} 1 & x_1 & x_1^2 & x_1^3 & ... \\ 1 & x_2 & x_2^2 & x_2^3 & ... \\ 1 & x_3 & x_3^2 & x_3^3 & ...\\ 1 & ...& ... & ... & ...\\ \end{bmatrix} , \]
R possui um pacote para automatizar essa transformação, matrixcalc, exemplificado no trecho de código abaixo:Agora basta aplicar a mesma relação matricial da equação (8.8), no caso, para quatro pontos intercalados dos dados experimentais acima, e portanto produzindo um polinômio de 4o grau:
T=c(5.29,15.23,25.11,37.39)+273 # dados de temperatura, em graus Kelvin
dG=c(11.74,12.46,12.88,13.13)*-1e3 #
library(matrixcalc)
# Criação das matrizes A (Vandermonde) e b
b=as.matrix(dG,nrow=4,byrow=TRUE) # vetor b
A = vandermonde.matrix(alpha=T,n=4); A # função para criar matriz de alternância (Vandermonde)## [,1] [,2] [,3] [,4]
## [1,] 1 278 77445 2e+07
## [2,] 1 288 83077 2e+07
## [3,] 1 298 88870 3e+07
## [4,] 1 310 96342 3e+07sol.vnd <- solve(A)%*%b; sol.vnd# coeficientes polinomiais (4o. grau)## [,1]
## [1,] 5e+05
## [2,] -5e+03
## [3,] 2e+01
## [4,] -2e-02polynom:library(polynom) # converte vetor de coeficientes em polinômio simbólico
p <- as.polynomial(sol.vnd)
p2 <- as.function(p) # permite converter o polinômio pra função curve
plot(T,dG)
curve(p2,from=273,to=315, add=TRUE) # curva polinomial suave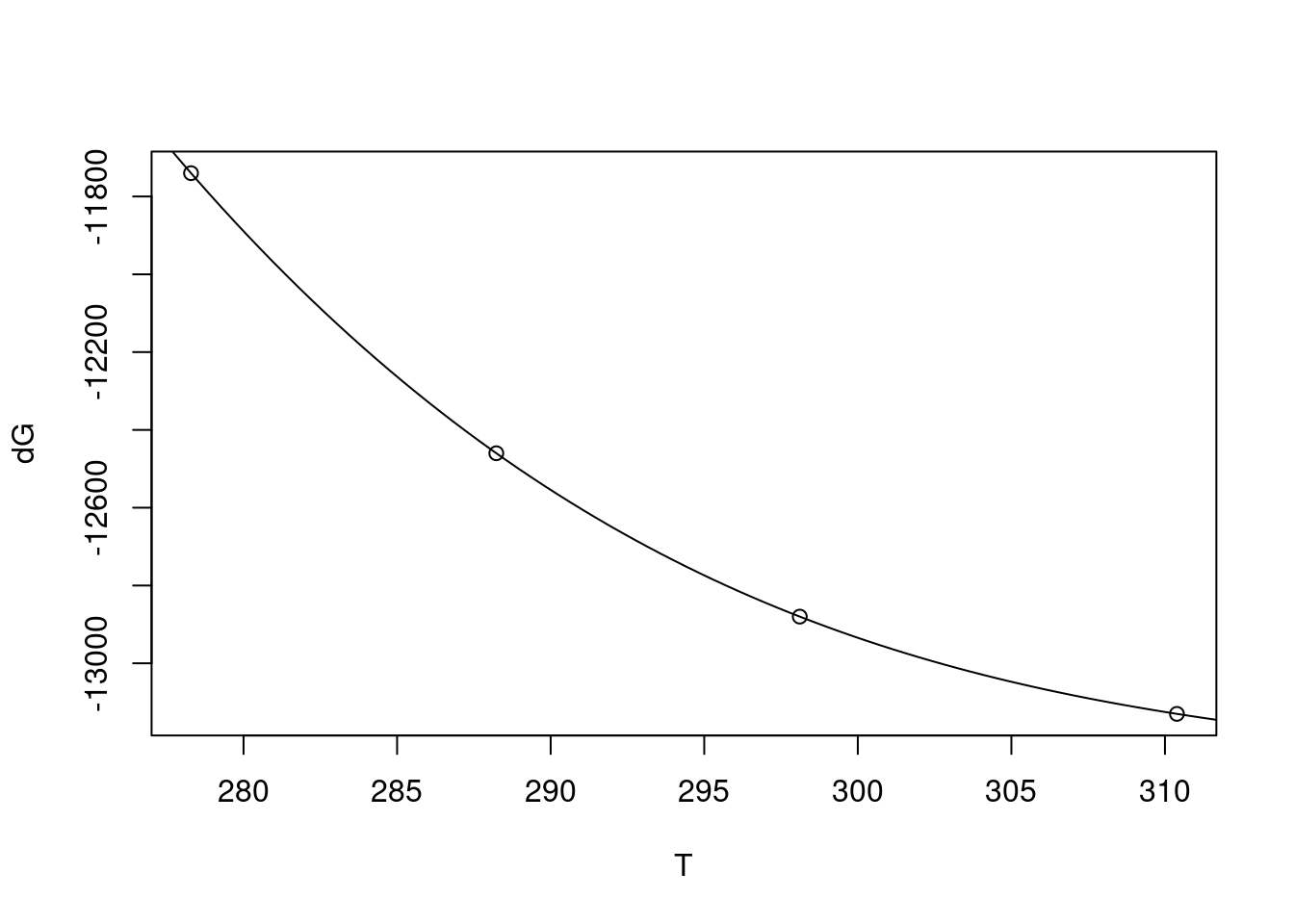
R (lm) como pela solução matricial acima, revele boa adesão do modelo aos dados experimentais, como representado pela Figura 8.2 e pela tabela de resultados acima, não há correlação de parâmetros termodinâmicos, posto tratar-se de um modelo matemático empírico, e não analítico para o sistema.Entretanto, é possível obter uma boa aproximação das quantidades \(\Delta\)H (entalpia), \(\Delta\)S (entropia) e \(\Delta\)Cp (capacidade calorífica) que modelam fenomenologicamente o comportamento termodinâmico em determinada temperatura, por relações próprias entre essas quantidades (Edelhoch and Osborne Jr 1976).
Assim, o valor de \(\Delta\)S pode ser obtido pela primeira derivada da relação empírica acima ((8.20)):
\[\begin{equation} \Delta S = -(\frac{\partial \Delta G}{\partial T})_p = -b-2cT-3dT^2 \tag{8.21} \end{equation}\]O valor de \(\Delta\)H, por sua vez, pode agora ser extraído da equação (8.1) aqui repetida, juntamente com a equação (8.20):
\[\begin{equation} \Delta G = \Delta H - T * \Delta S \tag{8.1} \end{equation}\] \[\begin{equation} \Delta H = \Delta G +T * \Delta S \tag{8.22} \end{equation}\]Ou seja, o valor de \(\Delta\)Cp pode ser aproximado pela primeira derivada de \(\Delta\)H (equação (8.23)) sobre T. Ou seja:
\[\begin{equation} \Delta Cp = -2cT-6dT^2 \tag{8.26} \end{equation}\]# Solução polinomial de parâmetros termodinâmicos para interação de insulina com receptor
T=c(5.29,10.07,15.23,20.21,25.11,30.29,37.39)+273 # dados de temperatura, em graus Kelvin
dG=c(11.74,12.17,12.46,12.73,12.88,12.98,13.13)*-1e3 # dados de -deltaG, em kcal/mol
# Ajuste a polinômio de 2o. grau
pol3 <- lm(dG~poly(T,3,raw=TRUE)) # ajuste a polinômio de 3o. grau
Tref = 298 # temperatura de referência, em graus Kelvin
# Cálculos
dG = coef(pol3)[1]+coef(pol3)[2]*Tref+coef(pol3)[3]*Tref^2+coef(pol3)[4]*Tref^3 # deltaG
dS = -coef(pol3)[2]-2*coef(pol3)[3]*Tref-3*coef(pol3)[4]*Tref^2 # deltaS
dH = coef(pol3)[1]-coef(pol3)[3]*Tref^2-2*coef(pol3)[4]*Tref^3 # deltaH
dCp = -2*coef(pol3)[3]*Tref-6*coef(pol3)[4]*Tref^2 # deltaCp
# Parâmetros em 298 K
cat("valor de deltaG: ",dG,"cal/mol","\n")## valor de deltaG: -12868 cal/molcat("Valor de deltaS: ",dS,"cal/mol/K","\n")## Valor de deltaS: 27 cal/mol/Kcat("Valor de deltaH: ",dH, "cal/mol","\n")## Valor de deltaH: -4735 cal/molcat("valor de deltaCp: ",dCp, "cal/mol/K","\n")## valor de deltaCp: -538 cal/mol/K8.6 Estabilidade Termodinâmica de Biopolímeros
O que resulta em:
\[\begin{equation} f_N = 1 - f_D \tag{8.28} \end{equation}\]Onde Si representa o sinal no ponto i.
E, portanto,
\[\begin{equation} \Delta G_D = -RT*ln\;K_D \tag{8.33} \end{equation}\]Tm=85
dHm=180
dCp=3
x=0:80
curve(dHm*(1-x/Tm)+dCp*((x-Tm-x*log(x/Tm))),xlim=c(0,80)) # Nicholson1996; Sholz2009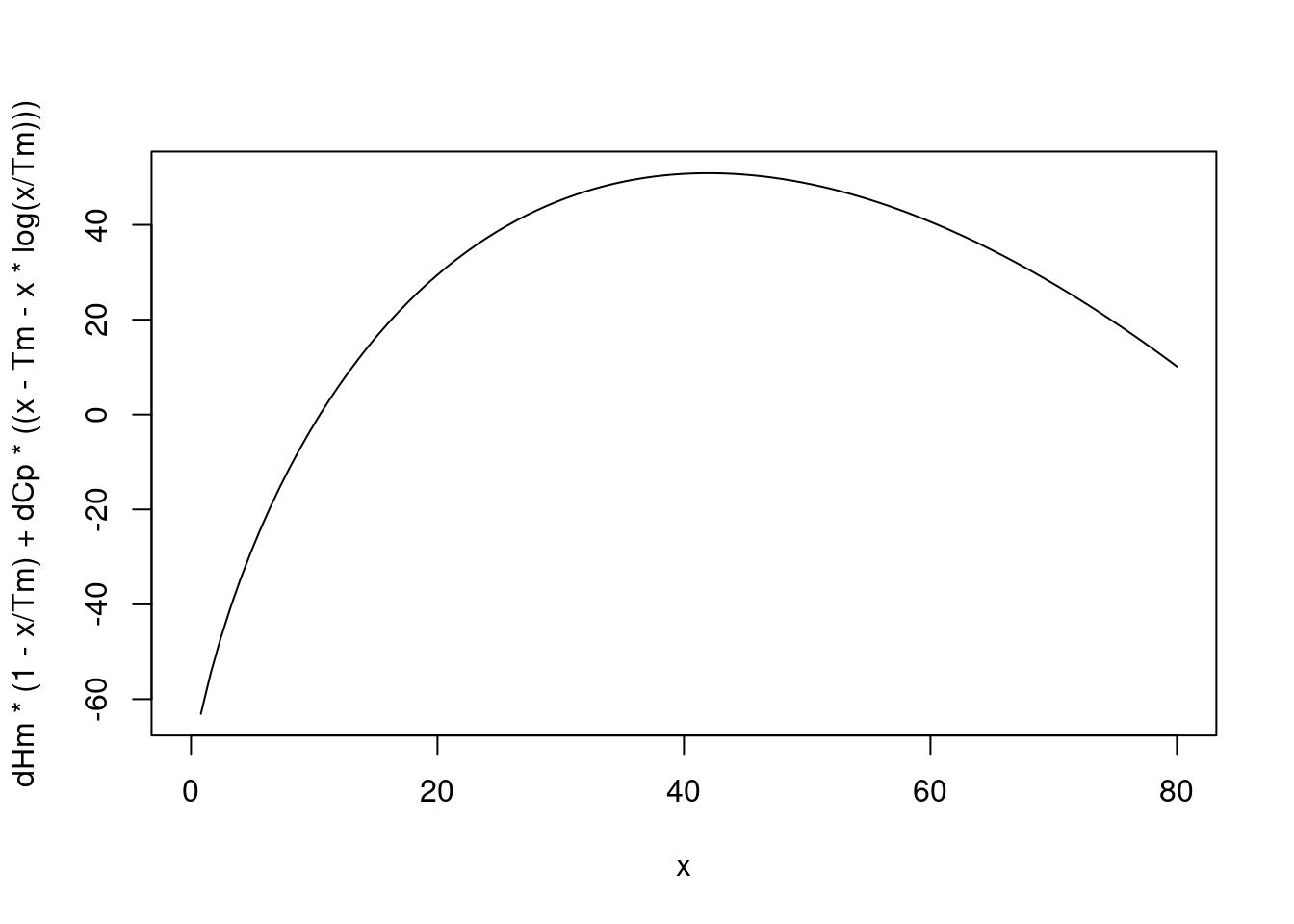
Figura 8.3: Curva de estabilidade simulada para a desnaturação de uma proteína. Tm = 75oC, DeltaHm = 180 kcal/mol, e DeltaCp = 3 kcal/mol/K.
8.7 Relação Quantitativa Estrutura-Função (QSAR) e Ajuste Multilinear
|Onde pIC\(_{50}\) representa a atividade biológica aferida (-log IC\(_{50}\)), S indexa valores de solubilidade, e W refere-se a parâmetro de largura do primeiro átomo do grupo substituinte. Esses dados são tabelados abaixo:
grupo <- c("H","Cl","SCH3","OCH3","CN","CHO","Br","CH3","CCH") # grupos substituintes em TIBO
S <- c(3.53,4.24,4.09,3.45,2.96,2.89,4.39,4.03,3.8) # parâmetro de solubilidade
W <- c(1,1.8,1.7,1.35,1.6,1.6,1.95,1.6,1.6) # parâmetro de largura de grupo
pIC50 <- c(7.36,8.37,8.3,7.47,7.25,6.73,8.52,7.87,7.53) # atividade biológica de TIBO
tab.tibo <-data.frame(grupo,S,W,pIC50)
knitr::kable(tab.tibo, caption="dados multivariáveis de atividade biológica de TIBO e parâmetros preditivos.", "pipe") # tabela| grupo | S | W | pIC50 |
|---|---|---|---|
| H | 4 | 1 | 7 |
| Cl | 4 | 2 | 8 |
| SCH3 | 4 | 2 | 8 |
| OCH3 | 4 | 1 | 8 |
| CN | 3 | 2 | 7 |
| CHO | 3 | 2 | 7 |
| Br | 4 | 2 | 8 |
| CH3 | 4 | 2 | 8 |
| CCH | 4 | 2 | 8 |
R permite fazê-lo por ao menos duas formas: função interna de ajuste linear (lm) ou álgebra matricial.8.7.1 Ajuste linear múltiplo por função lm:
lm.tibo <- lm(tab.tibo$pIC50~tab.tibo$S+tab.tibo$W) # comando para ajuste multilinear;
# Alternativamente,
# lm.tibo <- lm(cbind(S,W)~pIC50)
summary(lm.tibo)##
## Call:
## lm(formula = tab.tibo$pIC50 ~ tab.tibo$S + tab.tibo$W)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.2764 -0.1565 0.0292 0.0891 0.2476
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 3.590 0.543 6.61 0.00058 ***
## tab.tibo$S 0.957 0.152 6.30 0.00075 ***
## tab.tibo$W 0.362 0.302 1.20 0.27589
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.2 on 6 degrees of freedom
## Multiple R-squared: 0.912, Adjusted R-squared: 0.883
## F-statistic: 31.1 on 2 and 6 DF, p-value: 0.000683R (*dataframe $ vetor). Essa é maneira mais simples, pois independe de pacotes extras (como o dplyr), embora seja menos legível.8.7.2 Ajuste linear múltiplo por matrizes:
\[X = \begin{bmatrix} 1 & S_{1} & W_{1} \\ 1 & S_{2} & W_{2}\\ 1 & S_{3} & W_{3}\\ ... & ... \end{bmatrix} \]
# Criação das matrizes A e b
A=matrix(c(rep(1,9),S,W),nrow=9,byrow = FALSE) # cria matriz com valor unitário necessário antes da variável independente
b=as.matrix(pIC50,nrow=1,byrow=FALSE) # vetor b
# Solução matricial do ajuste linear
beta = solve(t(A) %*% A) %*% t(A) %*% b; beta## [,1]
## [1,] 3.6
## [2,] 1.0
## [3,] 0.4Esse procedimento matricial multilinear também pode ser aplicado em outros tipo de análise multivariável, como experimento fatorial e metodologia de superfície de resposta. Isso decorre da própria natureza desses sistemas, quando lineares. Veja as aplicações abaixo. Mesmo para metodologia de superfície de resposta quadrática (onde os parâmetros variam com o quadrado das variáveis preditoras), também é possível a solução matricial (equação (8.11)).
8.8 Rendimento de Reação & Planejamento Fatorial 2\(^{2}\)
# Dados do experimento
temp <-c(40,60,40,60)
catalis <-c("A","A","B","B")
rendim <- c(59,90,54,68)
# Tabela do planejamento fatorial
tab.fat <- data.frame(temp,catalis,rendim)
knitr::kable(tab.fat, caption="Dados de experimento fatorial (Neto e cols, 2010).", "pipe") # tabela| temp | catalis | rendim |
|---|---|---|
| 40 | A | 59 |
| 60 | A | 90 |
| 40 | B | 54 |
| 60 | B | 68 |
\[X = \begin{bmatrix} 1 & -1 & -1 & 1 \\ -1 & -1 & 1 & 1 \\ -1 & 1 & -1 & 1 \\ 1 & 1 & 1 & 1 \\ \end{bmatrix} \]
# Criação da matriz de planejamento codificada
X = matrix(c(1,-1,-1,1,1,1,-1,-1,1,-1,1,-1,1,1,1,1),nrow=4,byrow=TRUE)
# Criação da matriz de rendimento
Y = as.matrix(rendim)
# Determinação dos coeficientes beta:
beta = t(X) %*% Y/c(4,2,2,2); beta## [,1]
## [1,] 68
## [2,] 22
## [3,] -14
## [4,] -8\[\begin{equation} \begin{pmatrix} M \\ T \\ C \\ TC \\ \end{pmatrix} = \begin{pmatrix} 67,75 \\ 22,50 \\ -13,50 \\ -8,50 \\ \end{pmatrix} \tag{8.38} \end{equation}\]
Onde M representa a média global da resposta, T e C os efeitos principais (temperatura e catalisador), e TC o efeito da interação. Em síntese, os resultados sugerem que 1) a temperatura favoreceu o rendimento, especialmente para o catalisador A, 2) há redução do rendimento quando se substitui o catalisador A pelo B, e c) os maiores rendimentos são obtidos com o catalisador A na temperatura mais alta. Expressando-se esses resultados na função linear múltipla:
\[\begin{equation} y=67,75+22,5*T-13,5*C-8,5*T*C \tag{8.39} \end{equation}\]beta = (solve(t(X) %*% X) %*% t(X) %*% Y)*c(1,2,2,2); beta## [,1]
## [1,] 68
## [2,] 22
## [3,] -14
## [4,] -88.9 Metodologia de Superfície de Resposta (MSR)
Exemplificando para uma superfície de resposta linear (Neto, Scarminio, and Bruns 2010), num ensaio em que se deseja verificar o melhor rendimento (rend, %) de uma reação variando-se 3 níveis de concentração de reagente (conc, %) e três velocidades de agitação magnética (agit, rpm), pode-se como dantes elaborar a matriz de contrastes a partir das informações da tabela que segue:
conc <- c(45,55,45,55,50,50,50)
agit <- c(90,90,110,110,100,100,100)
x1 <- c(-1,1,-1,1,0,0,0)
x2 <- c(-1,-1,1,1,0,0,0)
rendim <- c(69,59,78,67,68,66,69)
tab.msr <- data.frame(conc,agit,x1,x2,rendim)
knitr::kable(tab.msr, caption="Dados de experimento de metodologia de superfície de resposta linear (Neto e cols, 2010).", "pipe") # tabela| conc | agit | x1 | x2 | rendim |
|---|---|---|---|---|
| 45 | 90 | -1 | -1 | 69 |
| 55 | 90 | 1 | -1 | 59 |
| 45 | 110 | -1 | 1 | 78 |
| 55 | 110 | 1 | 1 | 67 |
| 50 | 100 | 0 | 0 | 68 |
| 50 | 100 | 0 | 0 | 66 |
| 50 | 100 | 0 | 0 | 69 |
# Criação da matriz de coeficientes de contraste
X = matrix(c(1,1,1,1,1,1,1,-1,1,-1,1,0,0,0,-1,-1,1,1,0,0,0),nrow=7,byrow=FALSE)
# Criação da matriz de rendimento
Y = as.matrix(rendim)
# Determinação dos coeficientes beta:
beta = solve(t(X) %*% X) %*% t(X) %*% Y; beta## [,1]
## [1,] 68
## [2,] -5
## [3,] 48.9.1 Superfície Quadrática de Resposta
conc <- c(30,40,30,40,35,35,35,28,35,42,35)
agit <- c(115,115,135,135,125,125,125,125,139,125,119)
x1 <- c(-1,1,-1,1,0,0,0,-sqrt(2),0,sqrt(2),0)
x2 <- c(-1,-1,1,1,0,0,0,0,sqrt(2),0,-sqrt(2))
rendim <- c(86,85,78,84,90,88,89,81,80,86,87)
tab.msr2 <- data.frame(conc,agit,x1,x2,rendim)
knitr::kable(tab.msr2, caption="Dados de experimento de metodologia de superfície quadrática de resposta (Neto e cols, 2010).", "pipe") # tabela| conc | agit | x1 | x2 | rendim |
|---|---|---|---|---|
| 30 | 115 | -1 | -1 | 86 |
| 40 | 115 | 1 | -1 | 85 |
| 30 | 135 | -1 | 1 | 78 |
| 40 | 135 | 1 | 1 | 84 |
| 35 | 125 | 0 | 0 | 90 |
| 35 | 125 | 0 | 0 | 88 |
| 35 | 125 | 0 | 0 | 89 |
| 28 | 125 | -1 | 0 | 81 |
| 35 | 139 | 0 | 1 | 80 |
| 42 | 125 | 1 | 0 | 86 |
| 35 | 119 | 0 | -1 | 87 |
\[\begin{equation} \begin{pmatrix} 1 & -1 & -1 & 1 & 1 & 1 \\ 1 & 1 & -1 & 1 & 1 & -1 \\ 1 & -1 & 1 & 1 & 1 & -1 \\ 1 & 1 & 1 & 1 & 1 & 1 \\ 1 & 0 & 0 & 0 & 0 & 0 \\ 1 & 0 & 0 & 0 & 0 & 0 \\ 1 & 0 & 0 & 0 & 0 & 0 \\ 1 & -\sqrt(2) & 0 & 2 & 0 & 0 \\ 1 & 0 & \sqrt(2) & 0 & 2 & 0 \\ 1 & \sqrt(2) & 0 & 2 & 0 & 0 \\ 1 & 0 & -\sqrt(2) & 0 & 2 & 0 \\ \end{pmatrix} \tag{8.41} \end{equation}\]
# Criação da matriz de coeficientes de contraste para a superfície quadrática
X = matrix(c(rep(1,11),-1,1,-1,1,0,0,0,-sqrt(2),0,sqrt(2),0,-1,-1,1,1,0,0,0,0,sqrt(2),0,-sqrt(2),1,1,1,1,0,0,0,2,0,2,0,1,1,1,1,0,0,0,0,2,0,2,1,-1,-1,1,0,0,0,0,0,0,0),nrow=11,byrow=FALSE)
# Criação da matriz de rendimento
Y = as.matrix(rendim)
# Determinação dos coeficientes beta:
beta = solve(t(X) %*% X) %*% t(X) %*% Y; beta## [,1]
## [1,] 89
## [2,] 2
## [3,] -2
## [4,] -3
## [5,] -3
## [6,] 2R permite, por exemplo, a construção de um gráfico tridimensional que represente a função obtida, e sem a necessidade de pacote adicional: tal como segue:x <- seq(-1,1,0.1) # preditor x
y <- seq(-1,1,0.1) # preditor y
mrs <- function (x,y) {89.00+1.51*x-2.36*y-2.81*x^2-2.81*y^2+1.75*x*y} # função aplicada aos preditores
z <- outer(x,y,mrs) # saída do gráfico 3D (resposta)
res<-persp(x, y, z, xlab='x1', ylab='x2', zlab='z',shade=0.4,theta = 30, phi = 15,ticktype='detailed') # plotagem de superfície da função z(x,y)
pontos <- trans3d(x1,x2,rendim,pmat=res) # comando para adição de pontos experimentais
points(pontos,pch=19,col=1) # adição dos pontos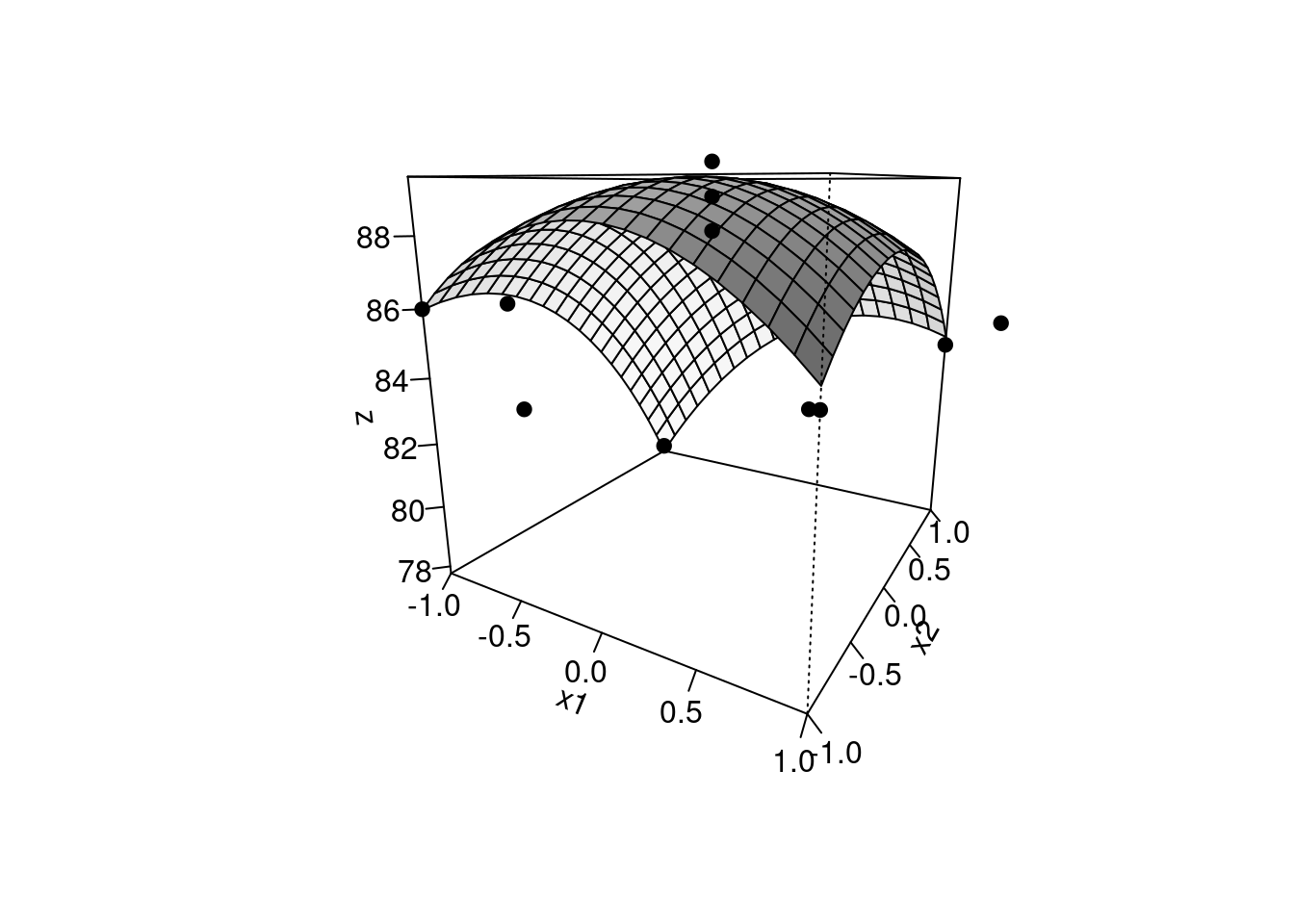
Figura 8.4: Superfície quadrática descrita pela equação de MSR com superposição dos valores experimentais.
R alguns pacotes para representação tridimensional de dados e funções (rgl, plot3D, scatterplot3d), como também para análise de planejamento fatorial (agricolae,afex,FMC), e de metodologia de superfície de resposta (rsm`).