Capítulo 7 Ácidos Nucleicos
7.1 Análise de sequências
Ácidos nucleicos podem ser considerados como sequências alfabéticas de 1 letra (bases), 2 letras (dinucleotídeo), ou de 3 letras (codon). Tomando-se o exemplo da lisozima de galinha:
- Acessa-se o banco de dados do NCBI - National Center for Biotechnology Information 9;
Seleciona-se o banco de dados Nucleotide;
Digita-se a sequência de interesse; ex: “hen egg” lysozyme”;
Seleciona-se LYZF1 (ou a referência de sequência do NCBI: NM_205281.2); Obs: Com o número de referência é possível acessar o conteúdo desejado a partir de uma consulta simples no Google.
Procura-se pela sequência referenciada em FASTA
O sítio apresentará a sequência nucleotídica para a lisozima, que pode ser copiada/colada no
R, ou exportada como arquivo em “Send to….File”.
Agora precisa-se converter esta sequência de letras (string) em um vetor de bases que possa ser lido pelo
R, e omitindo-se a quebra de linha. Isso pode ser agilizado com o pacote seqinr ou TmCalculator pela função s2c (converte string em vetor de strings; c2s faz o oposto). Ou também pelo pacote stringr:library(stringr)
liso.nucl <-"GCAGTCCCGCTGTGTGTACGACACTGGCAACATGAGGTCTTTGCTAATCTTGGTGCTTTGCTTCCTGCCC
CTGGCTGCTCTGGGGAAAGTCTTTGGACGATGTGAGCTGGCAGCGGCTATGAAGCGTCACGGACTTGATA
ACTATCGGGGATACAGCCTGGGAAACTGGGTGTGTGCCGCAAAATTCGAGAGTAACTTCAACACCCAGGC
TACAAACCGTAACACCGATGGGAGTACCGACTACGGAATCCTACAGATCAACAGCCGCTGGTGGTGCAAC
GATGGCAGGACCCCAGGCTCCAGGAACCTGTGCAACATCCCGTGCTCAGCCCTGCTGAGCTCAGACATAA
CAGCGAGCGTGAACTGCGCGAAGAAGATCGTCAGCGATGGAAACGGCATGAACGCGTGGGTCGCCTGGCG
CAACCGCTGCAAGGGCACCGACGTCCAGGCGTGGATCAGAGGCTGCCGGCTGTGAGGAGCTGCCGCGCCC
GGCCCGCCCGCTGCACAGCCGGCCGCTTTGCGAGCGCGACGCTACCCGCTTGGCAGTTTTAAACGCATCC
CTCATTAAAACGACTATACGCAAACGCC"
liso.nucl <- unlist(strsplit(liso.nucl,"")) # converte sequência gênica de uma palavra em nucleotídios separados
liso.nucl[1:100] # uma amostra do resultado## [1] "G" "C" "A" "G" "T" "C" "C" "C" "G" "C" "T" "G" "T" "G" "T"
## [16] "G" "T" "A" "C" "G" "A" "C" "A" "C" "T" "G" "G" "C" "A" "A"
## [31] "C" "A" "T" "G" "A" "G" "G" "T" "C" "T" "T" "T" "G" "C" "T"
## [46] "A" "A" "T" "C" "T" "T" "G" "G" "T" "G" "C" "T" "T" "T" "G"
## [61] "C" "T" "T" "C" "C" "T" "G" "C" "C" "C" "\n" "C" "T" "G" "G"
## [76] "C" "T" "G" "C" "T" "C" "T" "G" "G" "G" "G" "A" "A" "A" "G"
## [91] "T" "C" "T" "T" "T" "G" "G" "A" "C" "G"liso.nucl <- liso.nucl[liso.nucl !="\n"] # elimina a quebra de linha do resultado anterior
liso.nucl[1:100] # uma amostra do resultado sem os "\n"## [1] "G" "C" "A" "G" "T" "C" "C" "C" "G" "C" "T" "G" "T" "G" "T" "G" "T" "A"
## [19] "C" "G" "A" "C" "A" "C" "T" "G" "G" "C" "A" "A" "C" "A" "T" "G" "A" "G"
## [37] "G" "T" "C" "T" "T" "T" "G" "C" "T" "A" "A" "T" "C" "T" "T" "G" "G" "T"
## [55] "G" "C" "T" "T" "T" "G" "C" "T" "T" "C" "C" "T" "G" "C" "C" "C" "C" "T"
## [73] "G" "G" "C" "T" "G" "C" "T" "C" "T" "G" "G" "G" "G" "A" "A" "A" "G" "T"
## [91] "C" "T" "T" "T" "G" "G" "A" "C" "G" "A" Com a sequência gênica em mãos pode-se avaliar um extenso conjunto de propriedades ou manipular o vetor de bases, tal como referenciado em alguns pacotes do
R (seqinr, DNASeqtest, haplotypes, rDNAse). Também pode-se proceder algum manuseio mais simples para o gene selecionado, como abaixo:length(liso.nucl[liso.nucl == "A"]) # quantifica as bases de purina na sequência## [1] 133table(liso.nucl) # contagem de cada nucleotídio## liso.nucl
## A C G T
## 133 173 174 108library(seqinr)
liso.nucl2<-tolower(liso.nucl) # a biblioteca seqinr opera com letras minúsculas, havendo a necessidade de conversão das maiúsculas obtidas pelo FASTA
# seqinr::count(liso.nucl2,1) # a mesma operação acima, mas com a biblioteca seqinr, e outro formato de chamada
# seqinr:: count(liso.nucl2, 1 )
# seqinr::count(liso.nucl2,2) # teor de dinucleotídios
# seqinr::count(liso.nucl2,3) # teor de trinucleotídios Outras manipulações da sequência, como o conteúdo de pares GC, gráfico da sequência de dinucleotídios, conversão da sequência de bases em uma sequência numérica e sua plotagem, e obtenção da sequência de bases complementar, por exemplo, podem ser obtidos por:
nucls<-table(liso.nucl)
GC <-100 * (nucls[2]+nucls[3])/(nucls[1]+nucls[2]+nucls[3]+nucls[4])
cat("percentual de conteúdo GC em lisozima de galinha: ", round(GC,3))## percentual de conteúdo GC em lisozima de galinha: 59GC(liso.nucl) * 100 # o mesmo comando anterior, mas com a biblioteca seqinr## [1] 59# contag.liso <- count(liso.nucl2,2)
#
# barplot(sort(contag.liso)) # gráfico de barras do teor de dinucleotídios organizado por frequência
# Conversão de sequência nucleotídica em numérica
liso.nucl.numer <-gsub("T","4",gsub("G","3",
gsub("C","2",gsub("A","1",liso.nucl)))) # substitui bases por valores
liso.nucl.numer2<- as.numeric(liso.nucl.numer)
liso.nucl.numer2[1:100] # 100 primeiros valores da sequência## [1] 3 2 1 3 4 2 2 2 3 2 4 3 4 3 4 3 4 1 2 3 1 2 1 2 4 3 3 2 1 1 2 1 4 3 1 3 3
## [38] 4 2 4 4 4 3 2 4 1 1 4 2 4 4 3 3 4 3 2 4 4 4 3 2 4 4 2 2 4 3 2 2 2 2 4 3 3
## [75] 2 4 3 2 4 2 4 3 3 3 3 1 1 1 3 4 2 4 4 4 3 3 1 2 3 1# Obs: também pode ser obtido pelas funções s2n e n2s do pacote seqinr
seq.liso <- seq(1:length(liso.nucl))
plot(seq.liso,as.vector(liso.nucl.numer2),type="l",
xlim=c(100,300),main= "Sequência de bases entre resíduos 100 a 300",sub="A=1;C=2;G=3;T=4")
# Obtenção de sequência complementar
comp.liso.nucl <-seqinr::comp(liso.nucl)
head(seqinr::c2s(comp.liso.nucl),50) # apresenta os primeiros 50 nucleotídios complementares## [1] "cgtcagggcgacacacatgctgtgaccgttgtactccagaaacgattagaaccacgaaacgaaggacggggaccgacgagacccctttcagaaacctgctacactcgaccgtcgccgatacttcgcagtgcctgaactattgatagcccctatgtcggaccctttgacccacacacggcgttttaagctctcattgaagttgtgggtccgatgtttggcattgtggctaccctcatggctgatgccttaggatgtctagttgtcggcgaccaccacgttgctaccgtcctggggtccgaggtccttggacacgttgtagggcacgagtcgggacgactcgagtctgtattgtcgctcgcacttgacgcgcttcttctagcagtcgctacctttgccgtacttgcgcacccagcggaccgcgttggcgacgttcccgtggctgcaggtccgcacctagtctccgacggccgacactcctcgacggcgcgggccgggcgggcgacgtgtcggccggcgaaacgctcgcgctgcgatgggcgaaccgtcaaaatttgcgtagggagtaattttgctgatatgcgtttgcgg"7.2 Termoestabilidade de DNA
Em relação à propriedades físico-químicas de ácidos nucleicos, é bem conhecida a relação entre a termoestabilidade de DNA duplex e o conteúdo de pares GC, tal como explicitado pela relação empírica (Creighton et al. 2010):
Onde [Na] representa a concentração molar de sódio, f a fração de pares GC da sequência, L seu comprimento, e % o teor de formamida.
Dessa forma é possível prever o valor de Tm (“melting temperature”) que indexa a termoestabilidade de uma sequência polinucleotídica em função do teor salino. Ilustrando-se para uma comparação entre a sequência da lisozima de galinha e a humana (NCBI ref. NC_000012.12), na ausência de formamida:
Dessa forma é possível prever o valor de Tm (“melting temperature”) que indexa a termoestabilidade de uma sequência polinucleotídica em função do teor salino. Ilustrando-se para uma comparação entre a sequência da lisozima de galinha e a humana (NCBI ref. NC_000012.12), na ausência de formamida:
# Para lisozima de galinha:
gc.teor<-seqinr::GC(liso.nucl) # teor de pares GC da lisozima
Na.conc <-seq(0.005,0.2,0.001) # concentração de NaCl, mmol/L
Tm.Na<-(81.5+16.6*log10(Na.conc/(1+0.7*Na.conc))+41*gc.teor-500/length(liso.nucl)) # valor de Tm para a de galinha
# Para a lisozima humana
liso.nucl.h <-"AGCCTAGCACTCTGACCTAGCAGTCAACATGAAGGCTCTCATTGTTCTGGGGCTTGTCCTCCTTTCTGTT
ACGGTCCAGGGCAAGGTCTTTGAAAGGTGTGAGTTGGCCAGAACTCTGAAAAGATTGGGAATGGATGGCT
ACAGGGGAATCAGCCTAGCAAACTGTAAGTCTACTCTCCATAATTCCAGAGAATTAGCTACGTATGGAAC
AGACACTAGGAGAGAAGGAAGAAGAAGAAGGGGCTTTGAGTGAATAGATGTTTTATTTCTTTGTGGGTTT
GTATACTTACAATGGCTAAAAACATCAGTTTGGTTCTTTATAACCAGAGATACCCGATAAAGGAATACGG
GCATGGCAGGGGAAAATTCCATTCTAAGTAAAACAGGACCTGTTGTACTGTTCTAGTGCTAGGAAGTTTG
CTGGGTGCCTGAGATTCAATGGCACATGTAAGCTGACTGAAAGATACATTTGAGGACCTGGCAGAGCTCT
CTCAAGTCCTTGGTATGTGACTCCAGTTATTTCCCATTTTGAACTTGGGCTCTGAGAGCCTAGAGTGATG
CAGTATTTTTCTTGTCTTCAAGTCCCCTGCCGTGATGTGGGATTTTTATTTTTATTTTTATTTTATTTTA
TTTTATTTTTAAAGACAGTCTCACTGTGTGGCCCAGGCTGGAGTGCAGTGGCATGATCTCAGCTCACTGC
AACCTCTGCCTTCTGGGCTCAAGTGATTCTCGTGCTTCAGCCTTCTGAGTAGCTGTGACTACAGGTGTGT
ACCACCACACCCAGCTAATTTTTTGTATTTTCAGTACAGATGGGGTTTCACCATGTTGGCCAAGCTGGTC
TTGAACTCCTGGCCTCAAATGATCTGCCCACCTCAGCCTCCCAAAGTGGTAGGATTACAGGTGTGAACCA
CTGCACCCAGCCGACATGGGATTTTTAACAGTGATGTTTTTAAAGAATATATTGAATTCCCTACACAAGA
GCAGTAGGAACCTAGTTCCCTTCAGTCACTCTTTGTATAGGATCCCAGAAACTCAGCATGAAATGTTTTA
TTATTTTTATCTACTCTACTTGATTAACTATCTTTCATTTTCTCCCACACAATTCAAGATGTGCCATGAG
GAAAAGTTATTTTATAGTTTAGTACATAGTTGTCGATGTAATAATCTCTGTAGTTTTCAGATTGAATTCA
GACATTTCCCCTCAATAGCTATTTTTGAATGAATGAGTGAAGGGATGAAATCACGGAATAGTCTTGTTTT
CAAGATTCTAACTTGATATCCAAATTCACCTTTAGATATTATAAGAAAATTTCTATCAGAAAATCCTTAT
GTTTTTCTGATTAAAAAAAGCATTTTTCCATCAGCCTATGTATCTGCTATGAATTTACAAAATCTACTCA
ACAGCTCTGTTGATTTTTCTGTTCTTGGCTGAATGTTGCCTGAGGGATGGGAGCACGGGAAGGGTAAAAG
CAATGGAACAAACATGTATTTTAATATTTTAAAAGTATGTTATATTGTTCGTTGGTGTTACAAGATGATT
TGCATTACAAAAGGATTCTCTTACAAGTCCCTTATCTTAACACTAAAGTGCTAAGATATTTTATAAGTAA
ATCTTTATACTTATAAAACAAATCAGTAAAATAGAAGTAGCTAAGTAGAACTGATTTTGCTATAGAGTAT
AAGTCACTTAGTGTTGCTGTTTATTACTAAAAATAAGTTCTTTTCAGGGATGTGTTTGGCCAAATGGGAG
AGTGGTTACAACACACGAGCTACAAACTACAATGCTGGAGACAGAAGCACTGATTATGGGATATTTCAGA
TCAATAGCCGCTACTGGTGTAATGATGGCAAAACCCCAGGAGCAGTTAATGCCTGTCATTTATCCTGCAG
TGGTAAGACAAGCTAATATTTGACCAATCTGGTTATACTTACAAGAATTGAGACTCAATACAAATGAAAA
AGCCTTGAAAGGTTCATGAGGGACCTAGAAAAACTACATCTCAACTTCCAGAAAGTCATTATTATTTTCC
TCATAATTCCCTGAGTAAGAAATTAAAGAAGTGGTATCATAAAAGGTTGATGTTTTTTAATATACAGAAG
TTTCTGGAATGACCTATTAATTTACTGTCAATGGCCTTACTGATGCTTTGTCCAGAACAATGCCATTGCT
CCTGCTTACTTTGGGGAGGTTTTGGGATAATTTAGTTGTATGGTCCTTTTTCAATTGTTTTACTTTTTTT
TTTATGAAATGTTCTAAATGTATAGAAAATTAGAGACATTAGTATAATAAACAGCCATATGCCCATTATG
CACTTTAAAAGTTGTTAACATTTTGCCATAGTTGCTTCTTCTATGCCTTTTTTTTTTTTTTTTTTTTTTT
TTTTTTTTTGCTGAGAGTTTTTTGTTTGGTTTTGTTTTGTTTTATTTTGAGACAGGGTCTCCCTGTCCCC
AGGCTGTAGTGCAGTGGCACCATCACAGCTCACTGCAGCCTCAAGTGATCATCCCACCACAGCCTCCCAA
GTAGCTGGGACTACAGGTGTGCACCACCATGCCTGGCAAATTTTTGAAATTTTTAGTACAGGCAAATTCT
GTGTTGCCCAGGCTGGTCTTGAACTCCTGAGTTCAAGCAATCTTCCCACCTCAGCCTCCTTAAGTGCTGG
AATTACAGGCGTTAGCCACTGTACCTGGCTACTGCTGAGAGACTTTTAAGTGAATTAGGAACATGATGAT
ATTCCATTTCTAAATTCTTTAGTTTACATCTTCAAAAAATACAGTTCCTGTAGAATTATTATTGTAAATA
ACAAATTAACTTAAGGATTTATTTATTTGGAGTGAAACAAATATTTTACTGAACTCATAAAAATAGAAAT
ACCATGTGGAATCCTCAGTGTCAAAAATATTGCAGAAATCTTGCAAAGTTGATATTATTAAATTGTTAAA
TATTAAAATTCCCAATAAAGAACATTAATCTTATTTCTAAAATCCAGTTAATTAAAAAAATTTATATTAT
ATAATAATATTTGGTCATTAAATAAAAATTAGAAAATACAAATAAGAAAAATAACACCCATAATCTTACT
ACCCAGAGGTTTATAACCATGGGTAAATTCTGGTATATATTCTTCCAGAATGTATATCAATCATGTGTAT
GAATGTTAAATTATATCATACACATATAAACCCACATACAAACATGTAAATACTGTGTGCTTTTGCAAAA
ATTAAATTGTATTATACACACGGCTTTACAATTTGCTTCTTATCACACAAAATTATTTGCATGTCAGCAA
ATACAAATCGGTTTTTAATGATCTTTTGCTCCATTTTCCAGATGAGAAAAAAATACAAATCTGTATCATC
ATTTTAAAAGAATGACTAGAATTTTAATATATGAATATTCTATAATTTACTGATCCAATTGTTACTATTG
AGCACTTAGGTTGTTTCCATTTTTCCCTCATAAATTGCTATGAATAGCTTTTTGTATACATCTTTGGGTG
CATTTCTTATTTCTTTTGGATAAATTTTCAATAATAGAACTGCTGAGTAAAATATCACTAGGTGTTTTTT
TACAGTGTCTAGTGCAAAGAAGACCTTTAATCATTTTGTTAATACTTCCAGAGCTTCCAATGACTTTGGT
AAATGAAGAAAAAAATGCTTCATTTCATGCTGAATGGGAGAGAATGAAGAGAGTTTTCCCCAACAATTAC
ACATATATGGACTCATAGAAAATAATATCTTACCATTCTTTCCACAGCCTAACAGAAAAAAGCTGGCTAA
ACCTAAATTTAAAATAAAATATCTATTAAAGTTTTTATTCCTTACCACCTGTCTTTCAGCTTTGCTGCAA
GATAACATCGCTGATGCTGTAGCTTGTGCAAAGAGGGTTGTCCGTGATCCACAAGGCATTAGAGCATGGT
ATGTTTTAAGTGTTAAAAGGGAAAACTATCTTACTCTACTGTTGATATATACAATGAGAGCAGACTTTTA
AAGACCAAAGTATGCTAATGACACCTCAAAATTGCAGCTTTTGGCTTATGCTAAATGATGTATTACCTAC
ATCCTTGAAGAAACAATCTACTTTAACTGATCCAGAATCTTACTCTTTTACTCCTCAATTTATTTTAGGG
GATTTCTAGAGTTTTAAGATGCTTCACACTCTATCAGTTCCTTGTCATATCTTGAAATTCTTTTTAGAAT
AAGTAAGTGTGGGCCGGGCACAGTGGCTCACGCCTGTAATCCCAGCACTTTGGGAGACCGAGGCAGATGG
ATCACCTGAGGTCAGGAGTTCGAGACCAGCCTGCCTAACATGGCAAAACCCCATCTCCACTAAAAATACA
AAAAATTAGCTGGGTGTGGTGGCAGGTGCCTGTAATCCCAGCCACTCGGGAGGCTGAGGCAGGAGACTTG
CTTGAACCCGGGAGGTGGAGGTTGCAGAGGATTGCGCCATTGTACTTCAGCCTGGGCGACAGAGTGAGAC
TCTGTCTCAAATAAATACATAAAAAATAAATGTGGAATTCACTTTGCAGTTGCTGCTGTACAACGCACAT
TACTCAATCTTTATGTTCGGCATTCTATGCTCTACTGAGAAATTTGGGTAGGAGTGAAGTATTTTGTATA
CATATCTTCATTTAATAAATAGCAATAGCTGGGTCTATCTTACTATTTTATCTATTGATAAAATATTTTG
TTTCCCCAAGGAGTGCGAAGTATGTATATTACAATGAAGATATGTTTTAACCTTTCACCATTTGCTTCAT
CTTTTTCTACAGGGTGGCATGGAGAAATCGTTGTCAAAACAGAGATGTCCGTCAGTATGTTCAAGGTTGT
GGAGTGTAACTCCAGAATTTTCCTTCTTCAGCTCATTTTGTCTCTCTCACATTAAGGGAGTAGGAATTAA
GTGAAAGGTCACACTACCATTATTTCCCCTTCAAACAAATAATATTTTTACAGAAGCAGGAGCAAAATAT
GGCCTTTCTTCTAAGAGATATAATGTTCACTAATGTGGTTATTTTACATTAAGCCTACAACATTTTTCAG
TTTGCAAATAGAACTAATACTGGTGAAAATTTACCTAAAACCTTGGTTATCAAATACATCTCCAGTACAT
TCCGTTCTTTTTTTTTTTGAGACAGTCTCGCTCTGTCGCCCAGGCTGGAGTGCAGTGGCGCAATCTCGGC
TCACTGCAACCTCCACCTCCCGGGTTCACGCCATTCTCCTGCCTCAGCCTCCCGAGTAGCTGGGATTACG
GGCGCCCGCCACCACGCCCGGCTAATTTTTTGTATTTTTAGTAGAGACAGGGTTTCACCGTGTTAGCCAG
GATGGTCTCGATCTCCTGACCTTGTGATCCACCCACCTCGGCCTCCCAAAGTGCTGGGATTACAGGCGTG
AGCCACTGCGCCCGGCCACATTCAGTTCTTATCAAAGAAATAACCCAGACTTAATCTTGAATGATACGAT
TATGCCCAATATTAAGTAAAAAATATAAGAAAAGGTTATCTTAAATAGATCTTAGGCAAAATACCAGCTG
ATGAAGGCATCTGATGCCTTCATCTGTTCAGTCATCTCCAAAAACAGTAAAAATAACCACTTTTTGTTGG
GCAATATGAAATTTTTAAAGGAGTAGAATACCAAATGATAGAAACAGACTGCCTGAATTGAGAATTTTGA
TTTCTTAAAGTGTGTTTCTTTCTAAATTGCTGTTCCTTAATTTGATTAATTTAATTCATGTATTATGATT
AAATCTGAGGCAGATGAGCTTACAAGTATTGAAATAATTACTAATTAATCACAAATGTGAAGTTATGCAT
GATGTAAAAAATACAAACATTCTAATTAAAGGCTTTGCAACACA"
liso.nucl.h <- unlist(strsplit(liso.nucl.h,"")) # converte sequência gênica de uma palavra em nucleotídios separados
liso.nucl.h <- liso.nucl.h[liso.nucl.h !="\n"] # elimina a quebra de linha do resultado anterior
gc.teor.h<-seqinr::GC(liso.nucl.h) # teor de pares GC da lisozima hunana
Tm.Na.h<-(81.5+16.6*log10(Na.conc/(1+0.7*Na.conc))+41*gc.teor.h-500/length(liso.nucl.h)) # valor de Tm para a humana
# Curvas de simulação
plot(Na.conc,Tm.Na,type="l",col=2,
xlab="[Na+], M",ylab="Tm, oC")
lines(Na.conc,Tm.Na.h,type="l",col=3)
legend(x=0.15,y=75,legend=c("galinha","humana"),col=c(2,3),cex=1,lty=c(1,2))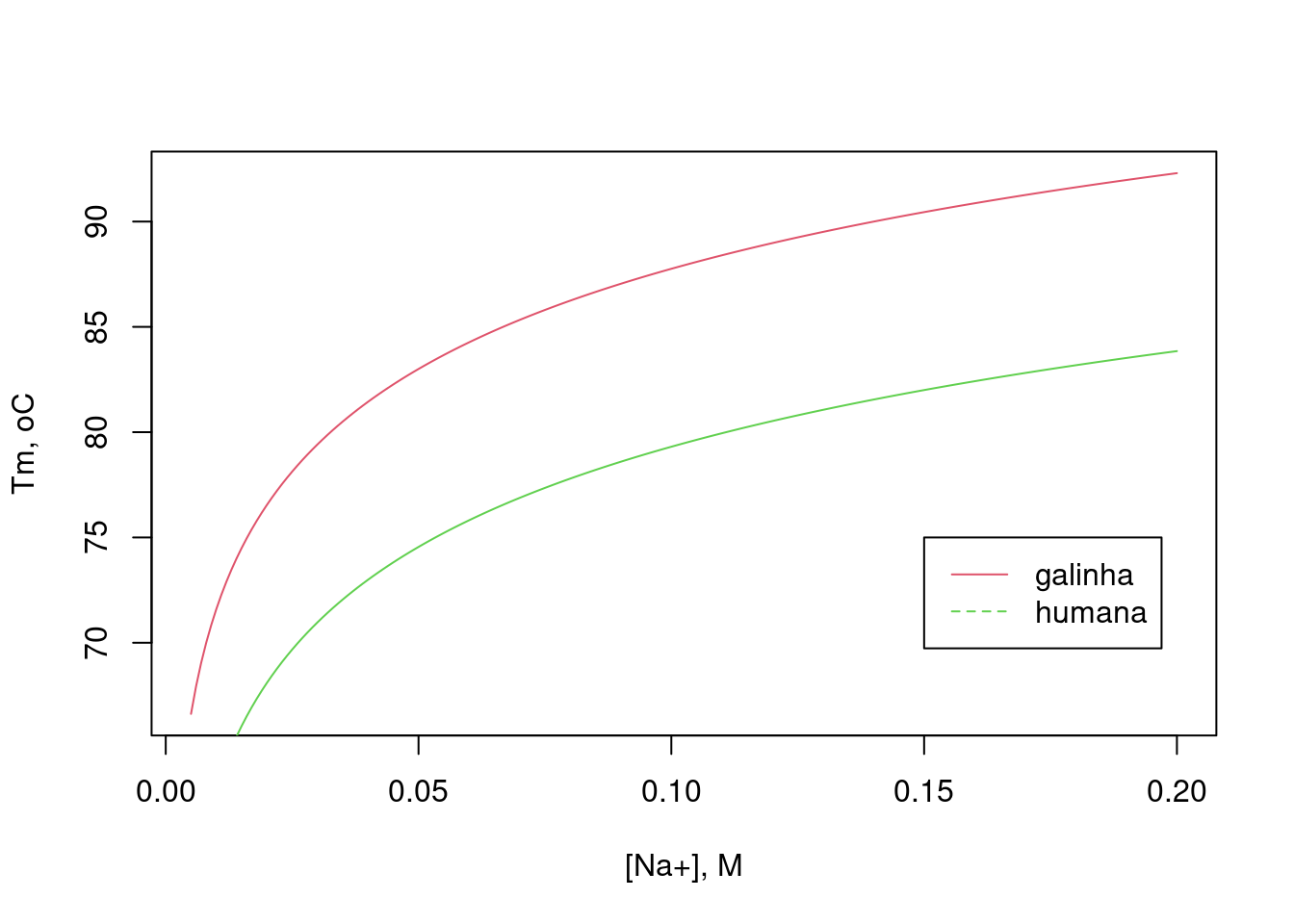
Figura 7.1: Comparação entre a curvas simuladas de Tm para a sequência nucleotídica da lisozima de galinha e lisozima humana, em função do teor de NaCl do meio.
Observe como a diferença no teor de GC tem efeito direto na termoestabilidade de fitas duplas de DNA. Uma observação: embora a faixa do valor de Tm relatado na literatura para a lisozima encontra-se em torno de 74C, esse valor refere-se à desnaturação cooperativa da enzima em solução aquosa, e não ao desenovelamento de sua sequência gênica de DNA duplex.
References
Creighton, Thomas E et al. 2010. Biophysical Chemistry of Nucleic Acids & Proteins. Distributed by Gardners Books.