# Argumentos para uma função
args(curve)function (expr, from = NULL, to = NULL, n = 101, add = FALSE,
type = "l", xname = "x", xlab = xname, ylab = NULL, log = NULL,
xlim = NULL, ...)
NULL\[\begin{equation} pH = pKa + log\frac{[A^-]}{[HA]} \label{hender-hassel} \end{equation}\]
\[\begin{equation} fa+fb=1 \label{frac-tit} \end{equation}\]
\[\begin{equation} pH = pKa + log\frac{fb}{1-fb} \label{HH-frac} \end{equation}\]
A partir dessa dedução, pode-se facilmente relacionar que:
\[\begin{equation} fb = \frac{10^{(pH-pKa)}} {1+10^{(pH-pKa)}} \label{HH-fb} \end{equation}\]
E, da mesma forma, pode-se encontrar fa como
\[\begin{equation} fa = 1- fb \label{HH-fb2} \end{equation}\]
Resultando em
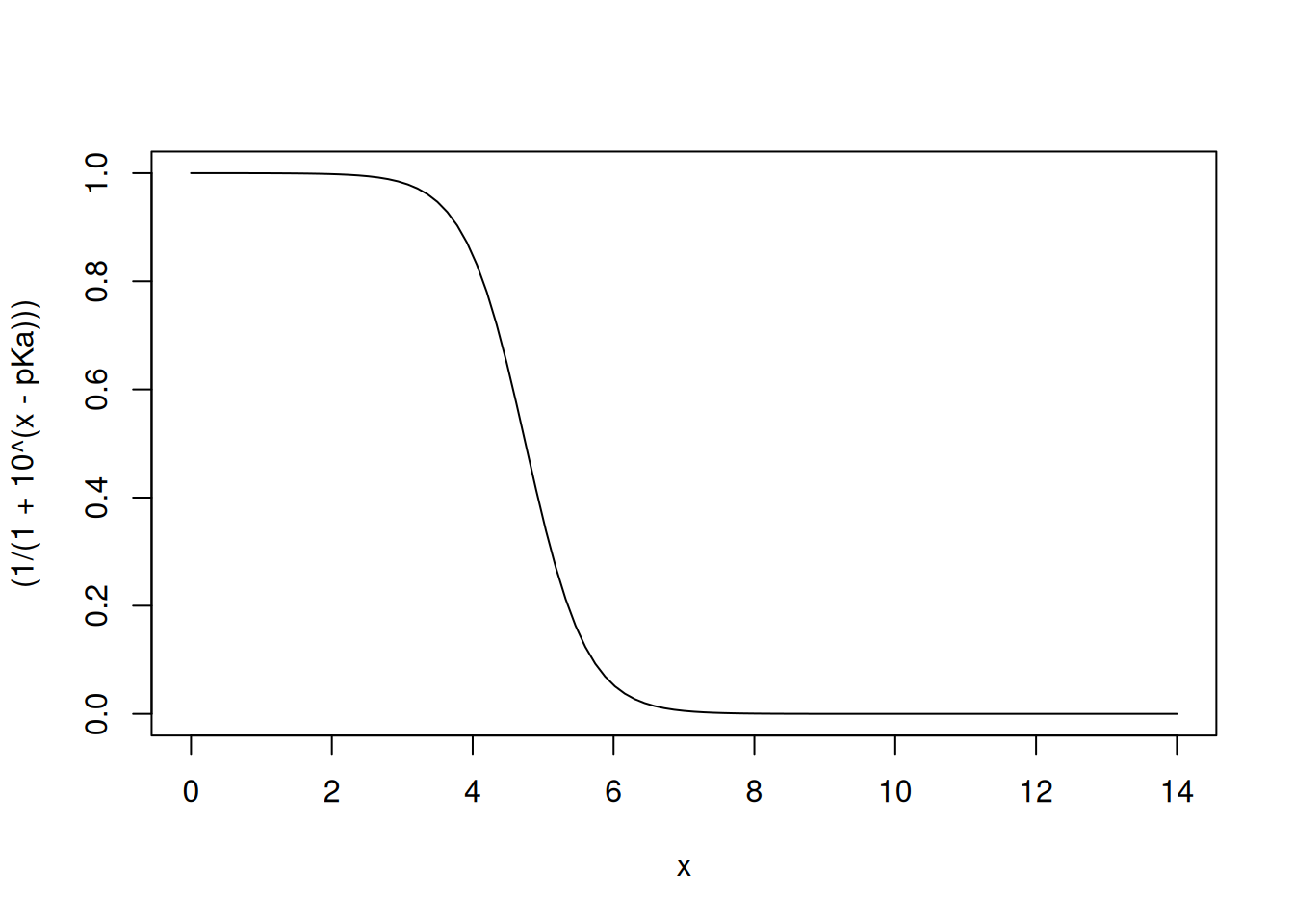
\[\begin{equation} fa = \frac{1}{1+10^{(pH-pKa)}} \label{eq-HH-fa} \end{equation}\]
curve a partir de seus argumentos (args), como segue:function (expr, from = NULL, to = NULL, n = 101, add = FALSE,
type = "l", xname = "x", xlab = xname, ylab = NULL, log = NULL,
xlim = NULL, ...)
NULLOu, de forma mais simples:
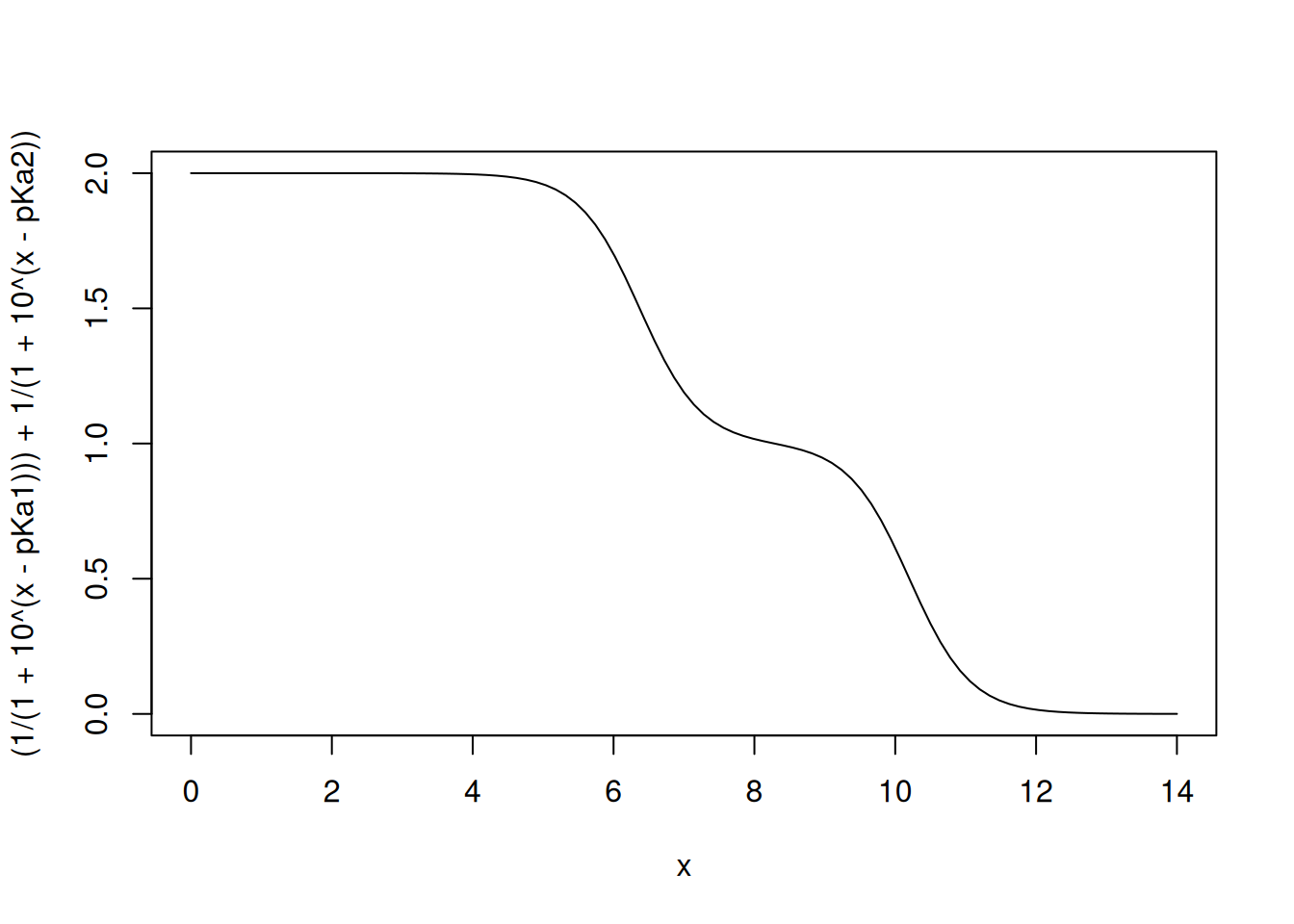
\[\begin{equation} fa = \frac{1}{1+10^{(pH-pKa1)}}+ \frac{1}{1+10^{(pH-pKa2)}} \label{HHbic} \end{equation}\]
Assim,
dev.copy:curve acima, e da flexibilidade que o pacote interno Graphics do R possibilita, pode-se elaborar uma curva mais complexa, como segue:pKa1 = 6.37
pKa2 = 10.20
curve((1/(1+10^(x-pKa1)))+1/(1+10^(x-pKa2)),0,14,
xlab="pH",ylab="fa",
main="Titulação de Ácido carbônico, H2CO3/HCO3-",
type="o", n=50,lwd=2,lty="dotted",
pch=3,col="blue",cex=1.2) # gráfico de titulação
text(4.7,1.3,"pKa = 6,37") # inserção de texto no gráfico
text(9,0.3,"pKa = 10,20")
abline(0.5,0, lty="dotted") # linha pontilhada em intercepto
# e inclinação específicos
abline(1.5,0, lty="dotted")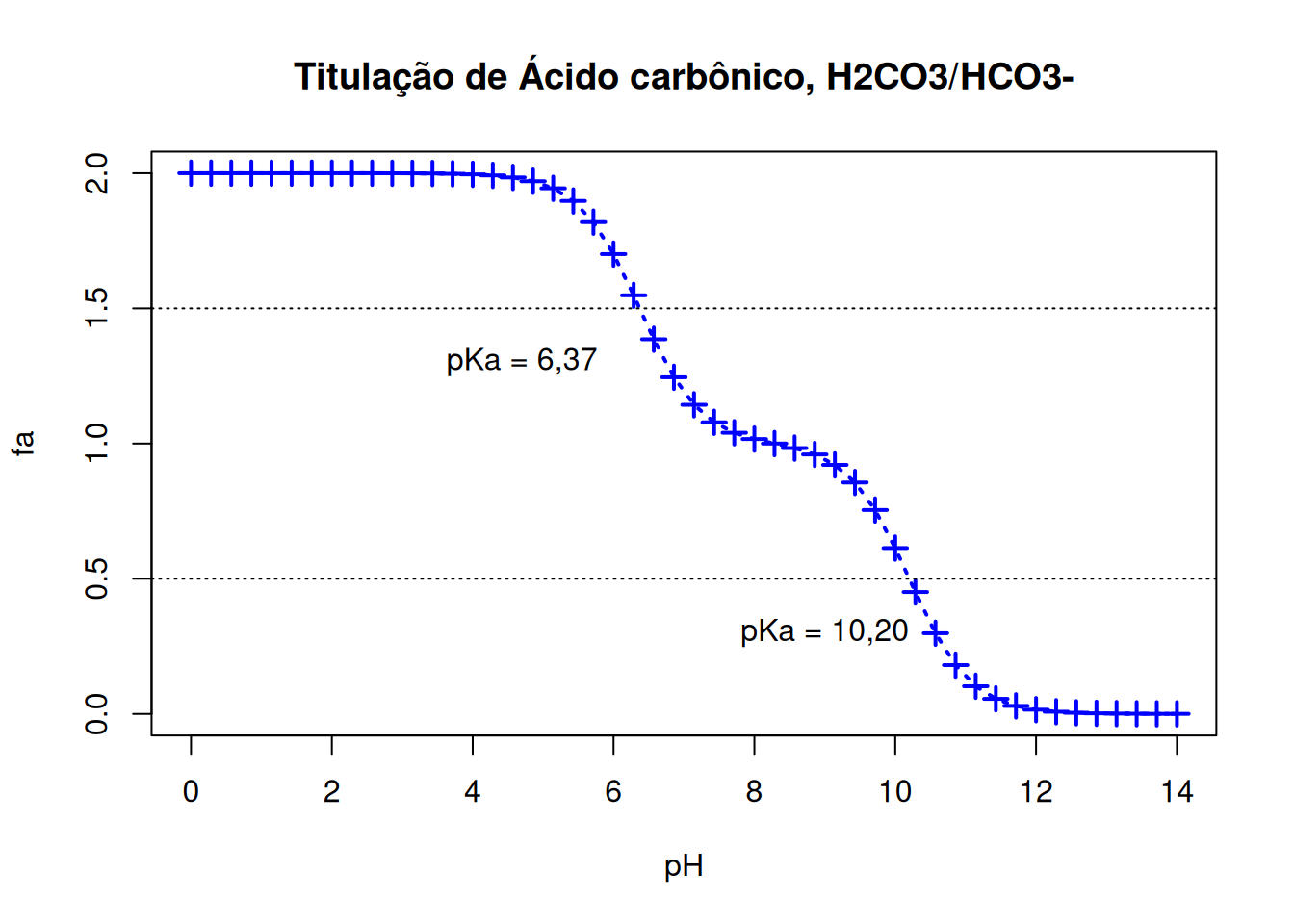
locator(). Como trata-se de um ponto apenas no gráfico, basta digitar o código locator(1) e clicar com o botão esquerdo do mouse no ponto da curva correpondente à fração de 0,5 para fa.\[\begin{equation} fa = \frac{1}{1+10^{(pH-pKa1)}}+ \frac{1}{1+10^{(pH-pK2)}}+\frac{1}{1+10^{(pH-pKa3)}} \label{eq-HHfosf} \end{equation}\]
No R isso pode ser feito como abaixo: \(\eqref{eq-HHfosf}\)
pKa1=2.2
pKa2=7.2
pKa3=12.7
curve((1/(1+10^(x-pKa1)))+
(1/(1+10^(x-pKa2)))+
(1/(1+10^(x-pKa3))),
xlim=c(1,14),
xlab="pH",ylab="fa",
main="Titulação de tampão fosfato",
sub = " As linhas pontilhadas cruzam os valores de pKa"
)
abline(v=c(2.2,7.2,12.7),col=c("blue","red","green"),lty="dotted") # adição de
# linhas verticais marcando os valores de pKa
text(1.6,2.5,"pKa1")
text(6.5,1.5,"pKa2")
text(11.8,0.5,"pKa3")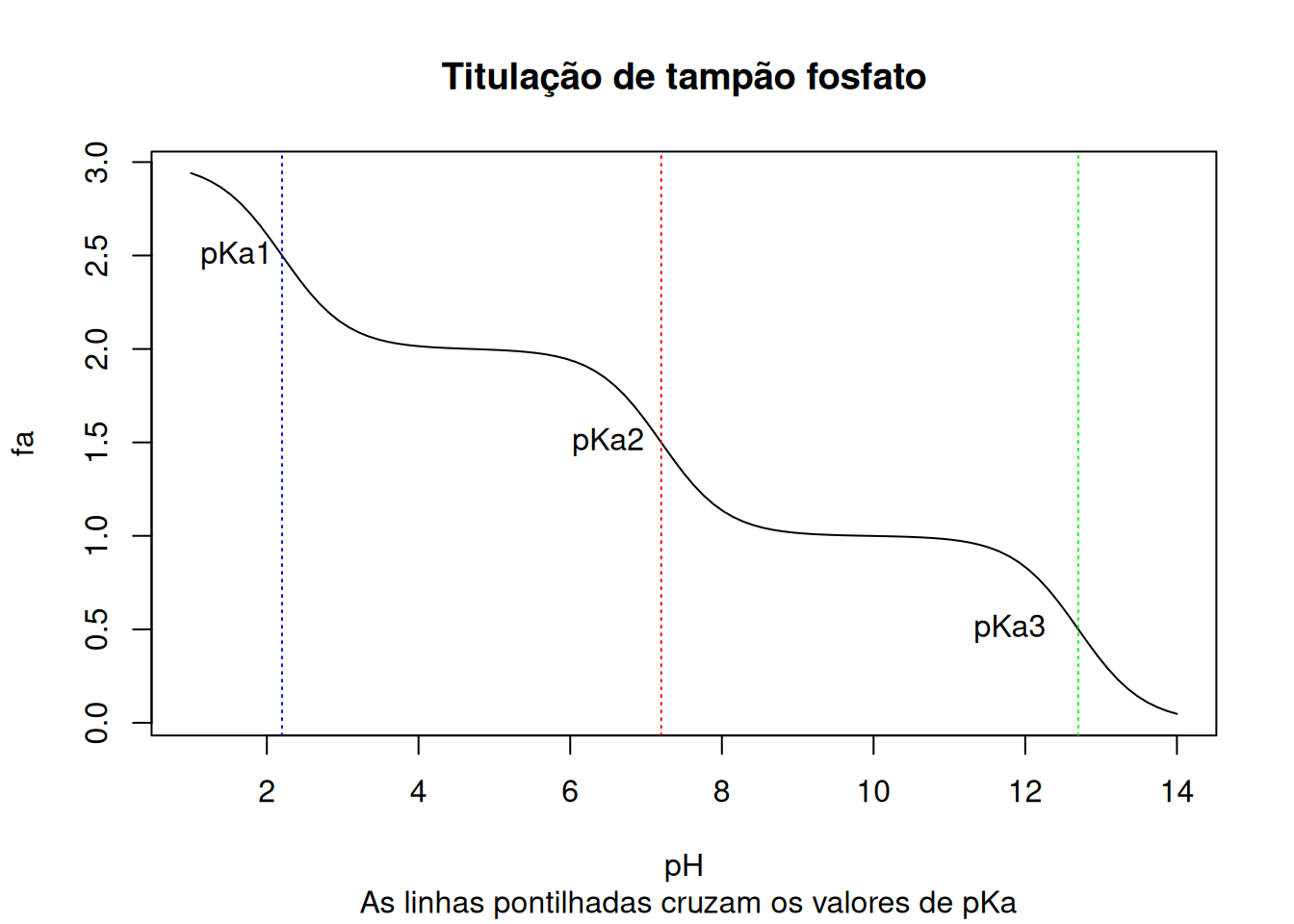
Definir uma função do R que contenha os parâmetros e a operação desejada.
Incluir na função uma estrutura de laço ou loop que permita repetir a operação até exaurido o número de prótons do composto.
Definir um vetor do R contendo os valores dos pKas do composto.
Definir a expressão de curva que viabilize a simulação.
Abaixo é apresentado um modelo de código que permite a simulação para o tampão fosfato.
Obs: o valor de pKa do sistema bicarbonato é de 6,8 quando considerado o \(CO_2\) como fonte de ácido carbônico \(H_2CO_3\) em sua reação com \(H_2O\), como por exemplo, para determinação de parâmetros arteriais em analisador hospitalar (\(CO_2\), \(HCO_3^-\), \(O_2\)).↩︎