1. Escolha um tema;
2. Clique no gráfico correspondente;
3. Clique em "add";
4. Use o mouse para interatividade e/ou edite o código.
Lembrete: o editor usa desfazer/refazer infinitos no código (Ctrl+Z / Shift+Ctrl+Z) !Simulações Interativas com JSPlotly
1 Simulações
1.1 Equilíbrio ácido-básico e sistema tampão
Contexto:
Equação:
\[
fa= \frac{1}{1 + 10^{pKa1 - pH}} + \frac{1}{1 + 10^{pKa2 - pH}} + \frac{1}{1 + 10^{pKa3 - pH}}
\] Onde, fa = fração de ácido (grupos protonáveis)
Sugestão:
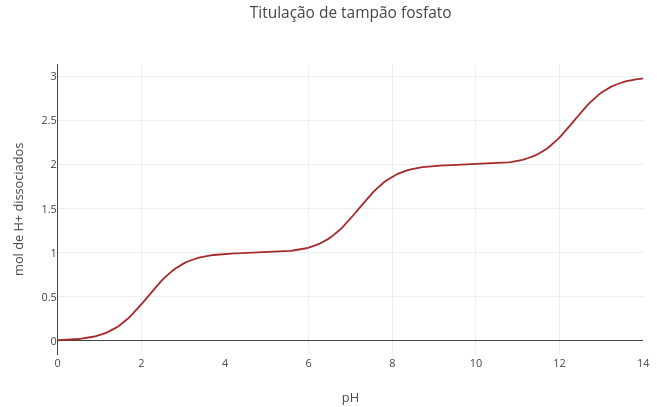
"A. Convertendo a curva de tampão fosfato (triprótico) para tampão bicarbonato (diprótico)"
1. Altere os valores de pKa para o tampão bicarbonato: pKa1 = 6.1, e pKa2 = 10.3;
2. Coloque um valor muito grande para pKa3 (ex:1e20).
3. Clique em "add plot".
Explicação: pKa é um termo que representa o logaritmo de uma constante de dissociação (-log[Ka]). Com um valor extremo, o denominador torna-se igualmente imenso, anulando o termo que leva pKa3. Em JavaScript e outras linguagens, "e" representa a notação para potência de 10.
"B. Convertendo a curva de tampão bicarbonato para acetato"
1. Basta repetir o procedimento acima, com pKa1 = 4.75, e eliminando-se pKa2.
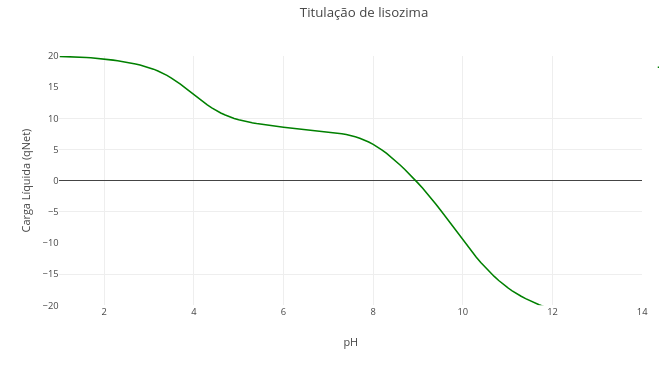
1.2 Estado de ionização de grupos químicos de biomoléculas
Contexto:
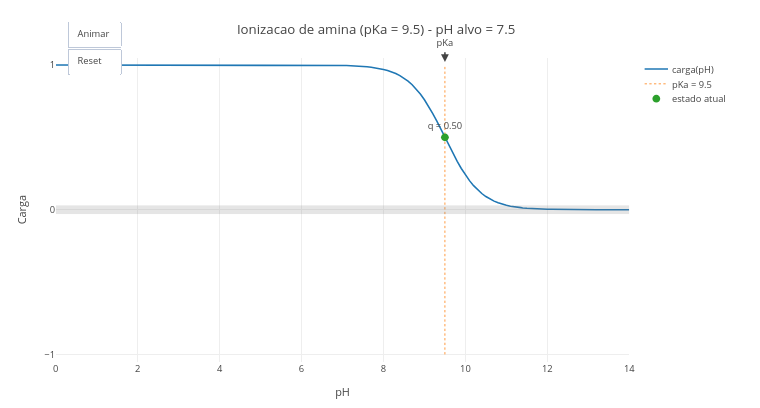
Instrução
Após clicar na imagem correlata abaixo, clique em “add” e observe o gráfico de titulação ácido-basica gerado. O eixo das abscissas (eixo x) possui uma faixa extensa de valores de pH, e o eixo das ordenadas a carga que a molécula com potencial de ionização se manifesta em cada pH, e em função de seu valor de pKa;
Clique em “Animar” para observar o deslocamento de uma bolinha ao longo da curva de titulação, até um valor de pH pré-definido no script do simulador, e em função de seu pKa;
Modifique no script o tipo de grupo ionizável (em “let grupo_edit =”) e/ou o pH final desejado (“let pH_final_edit =”), para avaliar o estado de ionização de um novo grupo;
Clique em “Reset” e “Animar”, para observar a nova animação.
Sugestão:
1. Troque o pH e o tipo de grupo que se pretende observar;
2. Simule o estado de ionização de moléculas diferentes, como fármacos com ácido carboxílico (ex: ácido acetilsalicílico...aspirina), no pH sanguíneo e na cavidade gástrica;
3. Crie um novo grupo ionizável e preveja seu estado de ionização. Para isso, basta alterar a informação em 3 constantes ao início do script: grupos, pKas, e tipos.
a.
1.3 Rede de carga líquida em peptídios
Contexto:
Equação:
\[
q =
\begin{cases}
-\dfrac{1}{1 + 10^{pK_a - pH}} & \text{(grupo ácido)} \\\\
\dfrac{1}{1 + 10^{pH - pK_a}} & \text{(grupo básico)}
\end{cases}
\]
Onde,
- pKa = valor do antilogarítmo de base 10 para a constante de equilíbrio de dissociação do ácido, Ka (ou log[Ka]).
Sugestão:
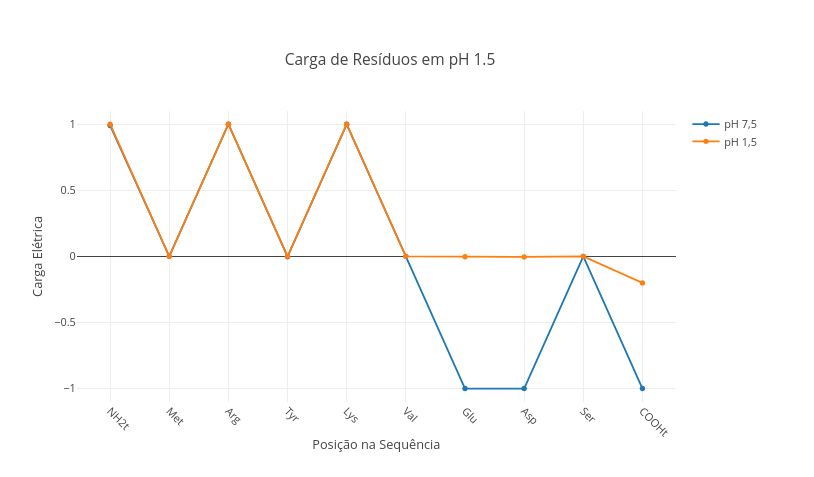
1. Selecione a sequência peptídica abaixo, e observe a distribuição de cargas:
"Ala,Lys,Arg,Leu,Phe,Glu,Cys,Asp,His"
2. Simule a condição de pH do estômago ("const pH = 1.5"), e verifique a alteração de cargas no peptídio.
3. Selecione um peptídio fisiológico (oxitocina, por ex), observe sua carga no sangue (pH 7.5), e reflita sobre seu potencial de interação eletrostática com componentes celulares.
"Cys,Tyr,Ile,Gln,Asn,Cys,Pro,Leu,Gly" - oxitocina
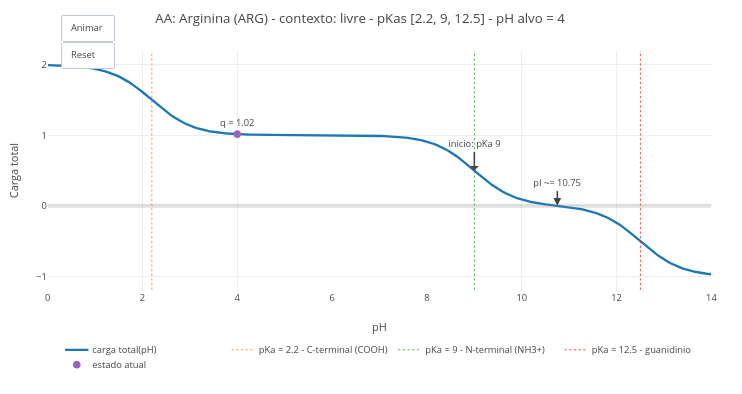
1.4 Carga, pH, pKa & pI de aminoácidos (animação)
Contexto:
Sugestão:
1. Simule a condição de um aminoácido no pH do sangue e no do estômago;
2. Compare os valores de pH de um aminoácido livre, com o de seu resíduo em cadeia;
3. Altere a opção booleana em "const showSites", para observar as curvas de cada grupo ionizável.
1.5 Ponto Isoelétrico em Proteínas
Contexto:
Equação:
\[
q_{\text{net}}(pH) = \sum_{i=1}^{N} \left[ n_i \cdot q_{B_i} + \frac{n_i}{1 + 10^{pH - pK_{a_i}}} \right]
\]
Onde,
- qnet = carga líquida total;
- qB$_{i} = carga da forma básica para o resíduo i (por exemplo, +1 para Lys, 0 para Asp);
- n\(_{i}\) = número de grupos do resíduo i.
Sugestão:
"Descobrindo o pI para outras proteínas"
1. Pode-se verificar a titulação de qualquer outra proteína ou sequência peptídica por simples substituição da sequência primária contida no código. Uma forma ascertiva de realizar essa substituição envolve:
a. Procurar a sequência "FASTA" da proteína no NCBI ("https://www.ncbi.nlm.nih.gov/protein/") - ex: "papain";
b. Clicar em "FASTA" e copiar a sequência 1a. obtida;
c. Colar a sequência num site para quantificação de resíduos (ex: "https://www.protpi.ch/Calculator/ProteinTool");
4. Substituir a sequência no código.
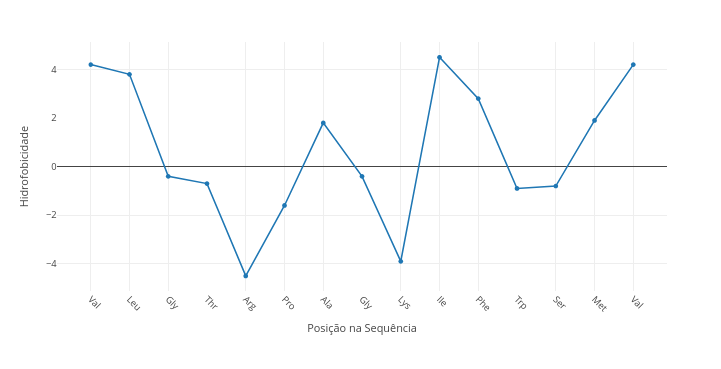
1.6 Hidrofobicidade em sequências de aminoácidos
Contexto:
Equação:
\[
\tilde{y}_i \;=\; \frac{1}{w} \sum_{k=i-m}^{i+m} y_k,
\qquad w = 2m+1
\]
Onde,
\[\begin{aligned} y_i &:\; \text{valor de hidrofobicidade na posição $i$ da sequência} \\ \tilde{y}_i &:\; \text{valor suavizado (média móvel) na posição $i$} \\ m &:\; \text{meia-janela (número de vizinhos de cada lado)} \\ w &:\; \text{largura total da janela ($w=2m+1$)} \\ i &:\; \text{posição do aminoácido na sequência ($1 \leq i \leq N$)} \end{aligned}\]Sugestão:
1. Experimente trocar a sequência para uma conhecida;
2. Compare uma sequência polar com uma apolar por sobreposição ("add");
3. Transcreva uma sequência de hélice transmembrânica de uma proteína acessando seus dados pelo site PDB
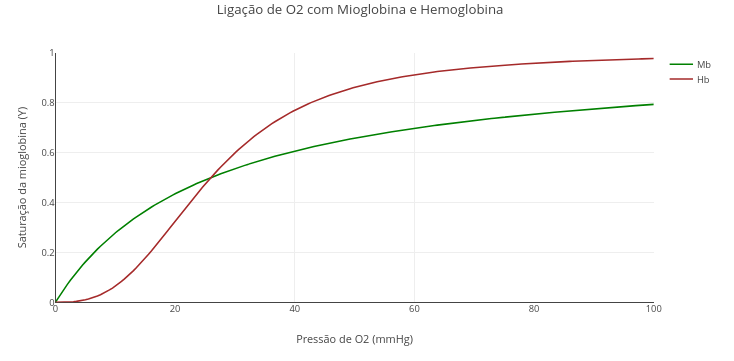
1.7 Interação de oxigênio com mioglobina e hemoglobina
Contexto:
Equação:
\[
Y= \frac{pO_2^{nH}}{p_{50}^{nH} + pO_2^{nH}}
\]
Onde
- Y = grau de saturação de oxigênio na proteína;
- pO\(_{2}\) = pressão de oxigênio;
- p\(_{50}\) = pressão de oxigênio a 50% de saturação;
- nH = coeficiente de Hill para a interação;
Sugestão:
1. Rode o aplicativo ("add plot"). Veja que o valor de "nH" da constante de Hill é "1", ou seja, sem efeito de cooperatividade.
2. Agora substitua o valor de "nH" pelo coeficiente de Hill para a hemoglobina, 2.8, e rode novamente !
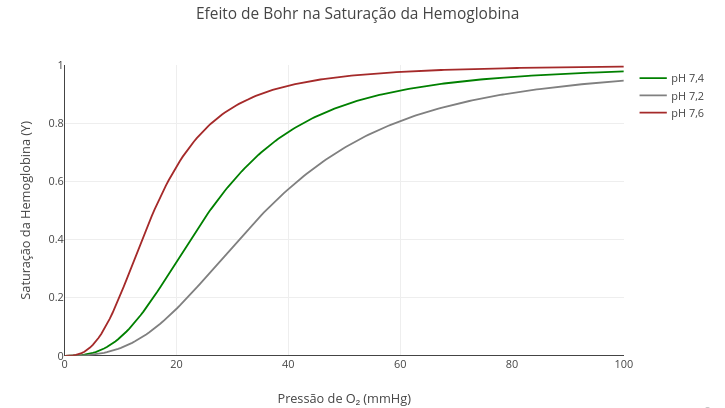
1.8 Efeito de Bohr em hemoglobina (pH)
Contexto:
Equação:
\[
Y(pO_2) = \frac{{pO_2^n}}{{P_{50}^n + pO_2^n}}, \quad \text{com } P_{50} = P_{50,\text{ref}} + \alpha (pH_{\text{ref}} - pH)
\]
Onde,
- Y = saturação da hemoglobina,
- pO\(_{2}\) = pressão parcial de oxigênio (em mmHg),
- P\(_{50}\) = pressão de O\(_{2}\) na qual a hemoglobina está 50% saturada,
- P\(_{50,ref}\) = 26 mmHg (valor padrão),
- \(\alpha\) = 50 (intensidade do efeito de Bohr),
- pH\(_{ref}\) = 7,4,
- n = 2,8 = coeficiente de Hill para a hemoglobina.
Sugestão
1. Experimente alterar o pH de referência para a interação;
2. Simule outros modelos alostéricos alterando o valor de "n"
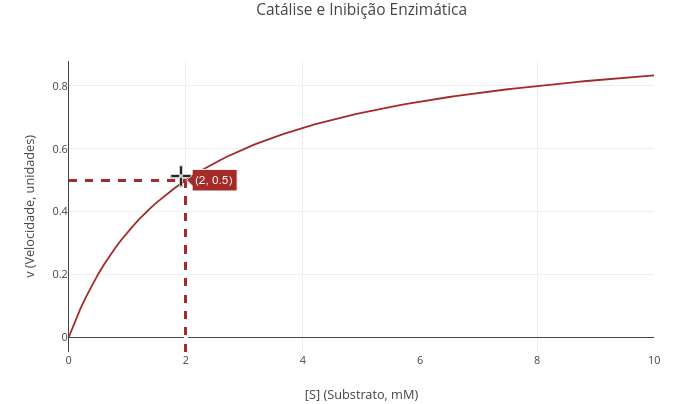
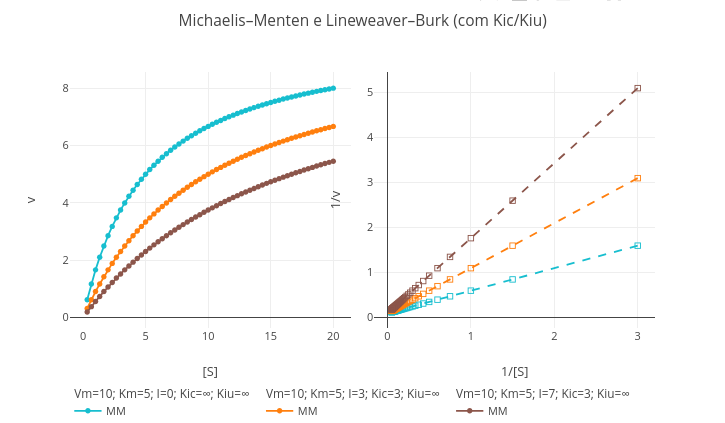
1.9 Catálise e inibição enzimática
Contexto:
Equação:
\[
v=\frac{Vm*S}{Km(1+\frac{I}{Kic})+S(1+\frac{I}{Kiu})}
\]
Onde
- S = teor de substrato para reação;
- Vm = velocidade limite da reação (nos livros, velocidade máxima);
- Km = constante de Michaelis-Mentem;
- Kic = constante de equilíbrio de dissociação de inibidor para modelo competitivo;
- Kiu = constante de equilíbrio de dissociação de inibidor para modelo incompetitivo
Sugestão:
"A. Catálise enzimática na ausência de inibidor."
1. Basta rodar o aplicativo com a equação geral. Veja que os valores para Kic e Kiu estão elevados (1e20). Dessa forma, com "constantes de dissociação" alta, a interação do inibidor com a enzima é irrelevante, retornando o modelo à equação clássica de Michaelis-Mentem.
2. Experimente alterar os valores de Vm e Km, comparando gráficos.
3. Use o recurso de coordenadas geográficas da barra de ícones ("Toggle Spike Lines"), para consolidar o significado matemático de Km, bem como observar o efeito de valores distintos desse sobre a visualização do gráfico.
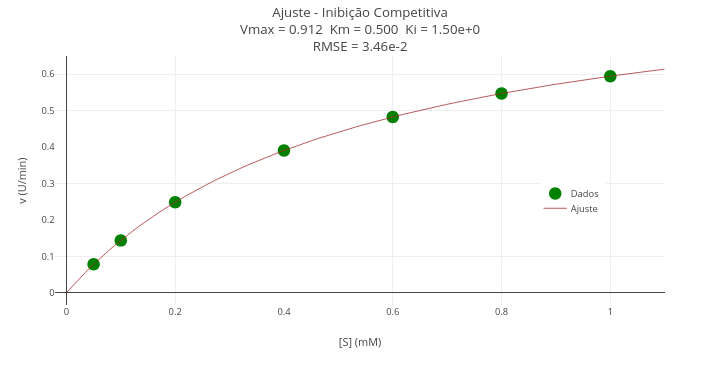
"B. Modelo de inibição competitiva."
1. Para observar ou comparar o modelo michaeliano com o de inibição competitiva, basta substituir o valor de Kic para um número consistente (ex: Kic= 3).
"C. Modelo de inibição incompetitiva."
1. A mesma sugestão acima serve para o modelo incompetitivo, desta vez substituindo o valor para Kiu.
"D. Modelo de inibição não competitiva pura."
1. Neste modelo, a simulação dá-se por valores iguais para Kic e Kiu.
"E. Modelo de inibição não competitiva mista."
1. Para este modelo, basta alocar valores distintos para Kic e Kiu.
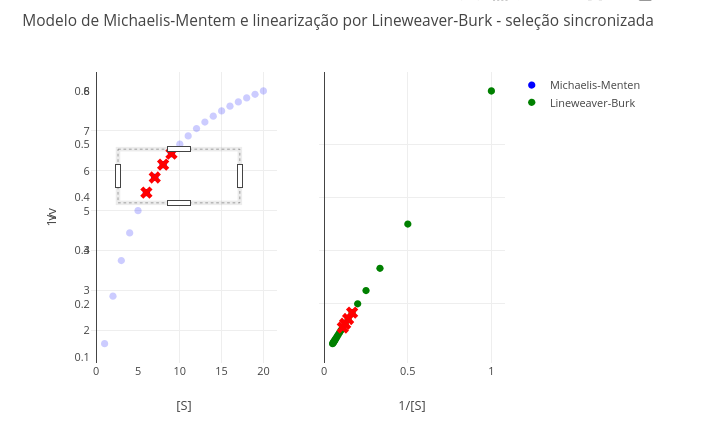
1.10 Linearização de dados cinéticos por Lineweaver-Burk
Contexto
1.10.1 Equação
\[
\frac{1}{v} = \frac{1}{S}*\frac{Km}{Vm} + \frac{1}{Vm}
\]
Sugestão
1. Experimente selecionar os extremos do gráfico de Michaelis-Mentem (curva hiperbólica), e observe os mesmos pontos no duplo-recíproco. Quais valores são mais confiáveis nesse último, os primeiros ou os últimos ?
2. Pesquise sobre outras formas de linearização (ex: Eadie-Hofstee; "v/S" X "v"), e veja como a transformação e seleção de pontos se apresenta. Para isso, altere as constantes abaixo:
const invS = S.map(s => 1/s);
const invV = V.map(v => 1/v);
#3# Diagnóstico de Inibição Enzimática (Michaelis-Mentem e Lineweaver-Burk)
Contexto
Sugestão
1. Simule as condições para uma inibição competitiva, inserindo um valor para o teor de inibidor ("I") e alterando o valor de "Km";
2. Faça o mesmo para um modelo incompetitivo, embora alterando o valor de "Vm";
3. Experimemente o modelo não competitivo puro, alterando "Km" e "Vm" para um mesmo valor;
4. Teste a inibição competitiva mista, inserindo valores distintos para "Km" e para "Vm."
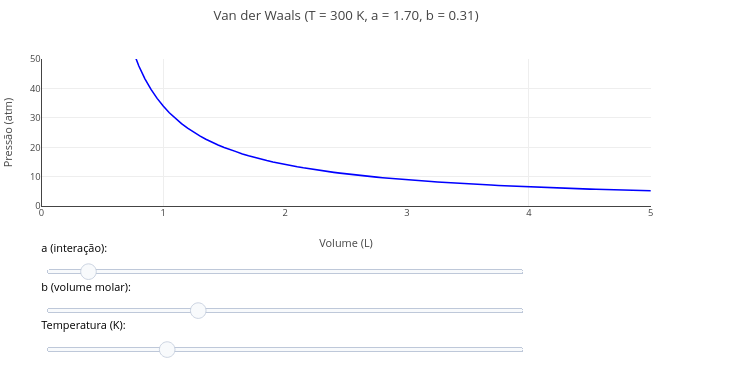
1.11 Equação de van der Waals para gases ideais
Contexto:
Equação:
\[ P = \frac{RT}{V - b} - \frac{a}{V^2} \]
- P = pressão do gás (atm);
- V = volume molar (L);
- T = temperatura (K);
- R = 0,0821 = constante dos gases ideais (L·atm/mol·K);
- a = constante de atração intermolecular (L\(^{2}\)·atm/mol$^{2})
- b = constante de volume excluído (L/mol)
Sugestão:
1. Experimente variar os parâmetros da equação por meio do "slider" para temperatura, bem como para os coeficientes "a e b".
2. Discorra sobre qual dos coeficientes possui maior efeito no perfil da curva, e a razão para isso.
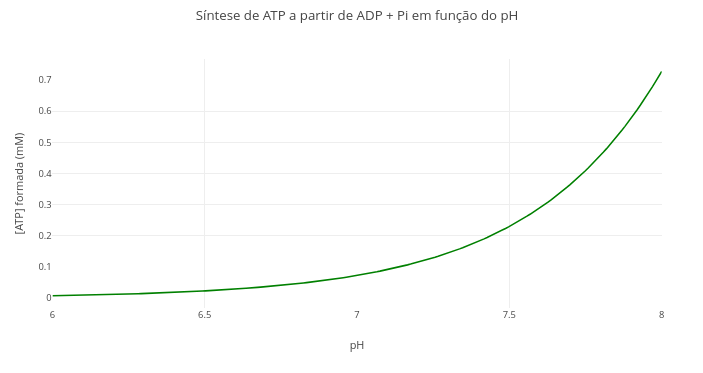
1.12 Equilíbrio de produção de ATP a partir de reagentes, temperatura, e pH
Contexto:
Equação:
\[ \Delta G = \Delta G^{\circ'} + RT \ln\left(\frac{[\text{ADP}] \cdot [\text{P}_i]}{[\text{ATP}]}\right) + 2{,}303 \cdot RT \cdot n_H \cdot \text{pH} \]
Onde,
- \(\Delta\)G = energia de Gibbs da reação (positivo para síntese espontaneamente desfavorável, kJ/mol);
- \(\Delta\)G\(^{o'}\) = 30,5 kJ/mol energia de Gibbs padrão biológica para a síntese de ATP;
- R = 8,314 J/mol/K (constante geral dos gases);
- T=310 K (temperatura fisiológica);
- nH\(^{+}\) = 1 (número de prótons envolvidos na reação);
- [ADP], [Pi], [ATP] = concentrações molares de reagentes e produto
Sugestão:
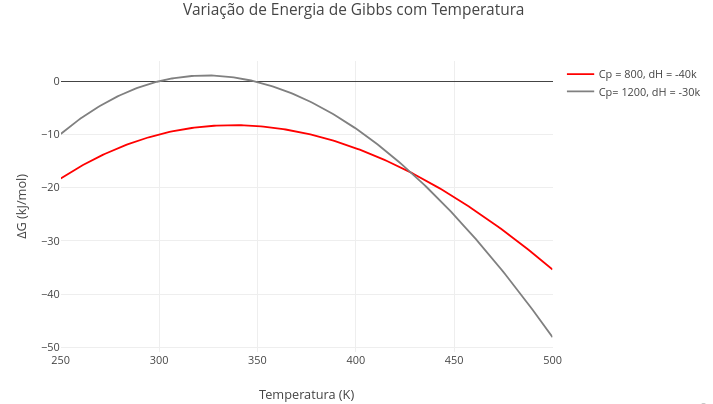
1. Altere as quantidades envolvidas na expressão, e compare com visualizações precedentes. Exemplificando, temperatura, pH, e teores de ADP e Pi.1.13 Variação da energia de Gibbs com a temperatura
Contexto:
Equação:
\[ \Delta G(T) = \Delta H^\circ - T\,\Delta S^\circ + \Delta C_p \left(T - T_0 - T \ln\left(\frac{T}{T_0}\right)\right) \]
Onde,
- \(\Delta\)G(T) = energia de Gibbs da reação em cada valor de temperatura, kJ/mol);
- \(\Delta\)H\(^{o}\) = entalpia padrão da reação a T\(_{0}\), normalmente 298 K (J/mol);
- \(\Delta\)S\(^{o}\) = entropia padrão da reação a T\(_{0}\);
- \(\Delta\)Cp = variação da capacidade calorífica da reação (J/mol·K), assumida constante com a temperatura;
- T = temperatura de interesse (K);
- T\(_{0}\) = temperatura de referência, geralmente 298 K.
- R = 8,314 J/mol/K (constante geral dos gases);
Sugestão:
1. Experimente variar um ou mais parâmetros da expressão;
2. Teste o comportamento da curva de Gibbs em uma temperatura de referência elevada (simulação para organismo extremófilo);
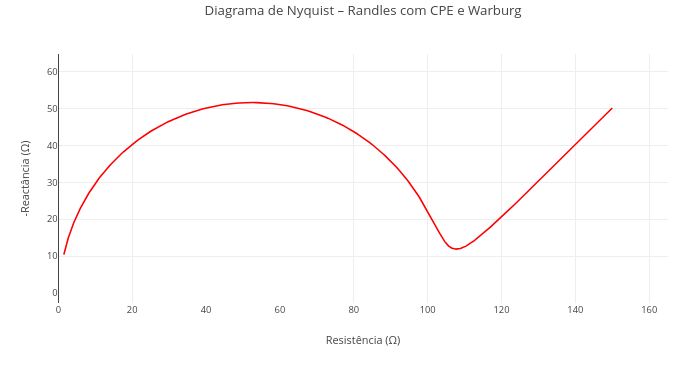
3. Simule a situação onde a variação da capacidade calorífica é nula1.14 Espectroscopia de impedância eletroquímica (EIS)
Contexto:
Equação:
\[ Z_{\text{total}}(\omega) = R_s + \left[ \left( \frac{1}{R_p} + Q (j\omega)^n \right)^{-1} \right] + \frac{\sigma}{\sqrt{\omega}} (1 - j) \]
Onde:
- Z\(_{total}\) = impedância total em determinada frequência ;
- \(\omega\) = Frequência angular (rad/s), \(\omega\)=2πf ou \(\omega\)=2πf;
- R\(_{p}\) = Resistência ôhmica (resistência da solução eletrolítica, fios, contatos etc.) ;
- R\(_{p}\) = Resistência de polarização (associada a processos de transferência de carga, como reações eletroquímicas);
- Q = Constante associada ao elemento de fase constante (CPE), substitui um capacitor ideal para representar comportamentos não ideais ;
- n = Expoente do CPE, entre 0 e 1; define o grau de idealidade do comportamento capacitivo (n = 1: capacitor ideal; n < 1: dispersão);
- \(\sigma\) = Coeficiente de Warburg, associado à difusão de íons no sistema eletroquímico ;
- j = Unidade imaginária, j\(^{2}\) = −1;
Sugestão
1. Verifique o efeito de Rs no gráfico, anulando o seu valor (resistência da solução);
2. Observe a deformação do semicírculo variando os valores do elemento de fase constante (ex: Q = 1e-3; n = 0.6 - dispersão do comportamento capacitivo);
2. Experimente combinar outros valores dos parâmetros do cabeçalho do código, para evidenciar situações correntes em eletroanálise: Rs, Rp, Q, n, e sigma;
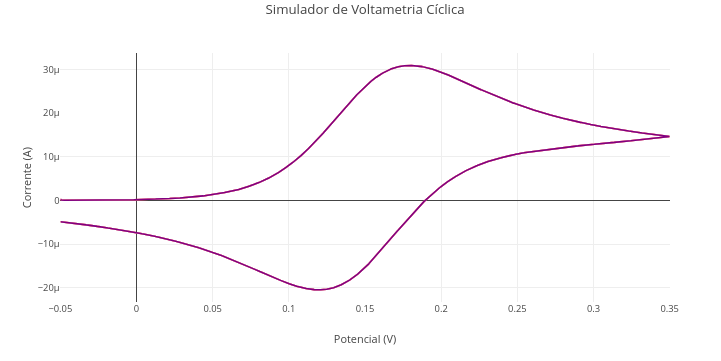
3. Reduza o modelo de Warburg com elemento de fase constante a um modelo de Randles simples, e formado apenas por 2 resistores em série, o segundo em paralelo com um capacitor ideal, e sem difusão de Warburg (sigma = 0; n = 1).1.15 Voltametria cíclica
Contexto:
Equação:
\[ j = j_0 \left[ \exp\left(\frac{\alpha n F (E - E^0)}{RT}\right) - \exp\left(\frac{-(1 - \alpha) n F (E - E^0)}{RT}\right) \right] \] Onde:
- j = densidade de corrente;
- j\(_{0}\) = corrente de troca;
- \(\alpha\) = coeficiente de transferência de carga;
- E = potencial aplicado ao eletrodo;
- E\(^{0}\) = potencial padrão do eletrodo;
- n = número de elétrons;
- F = constante de Faraday (96485 C·mol⁻¹)
- R = constante geral dos gases (8.314 J·mol⁻¹·K⁻¹);
- T = temperatura
\[ \frac{\partial C(x,t)}{\partial t} = D \frac{\partial^2 C(x,t)}{\partial x^2} \]
Onde:
- C(x,t) = concentração da espécie eletroanalítica (mol/cm³), em função da posição x e do tempo t;
- D = coeficiente de difusão da espécie (cm²/s);
- t = tempo (s);
- x = distância da superfície do eletrodo (cm);
- \(\delta\) = notação para derivada parcial.
\[ E = E^0 + \frac{RT}{nF} \ln \left( \frac{[\text{Ox}]}{[\text{Red}]} \right) \]
\[ i(t) = n F A D \left. \frac{\partial C(x,t)}{\partial x} \right|_{x=0} \] Onde:
- A = área de superfície eletródica (cm²);
- i(t) = corrente no tempo t (A, Àmpere).
Simulador de Voltametria Cíclica
Sugestão:
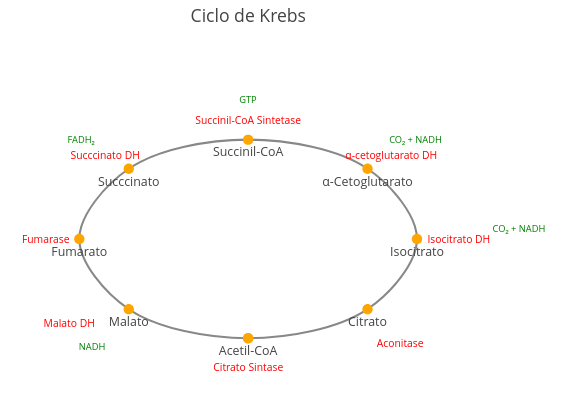
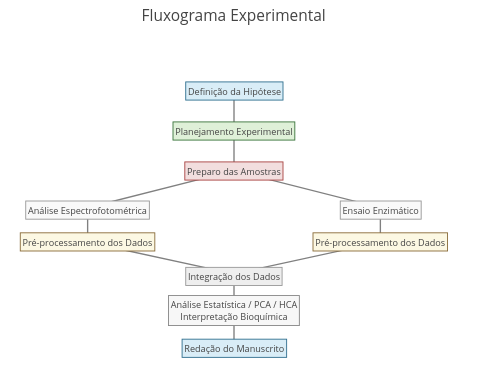
1. Observe o quantidade de valores ao início do código, tangíveis a uma "manipulação paramétrica". Procure saber o que representam, e busque variar seus valores de modo consciente, objetivando agregar valor ao aprendizado da simulação. Essa é a alma da "manipulação paramétrica" que envolve o "ensino reprodutível" !!1.16 Diagramas e fluxogramas
Contexto: Diagrama
Sugestão:
1. Experimente reposicionar melhor enzimas e metabólitos, apenas clicando e arrastando os termos;
2. Experimente substituir os nomes que estão no código para a produção de outro ciclo metabólico, como o ciclo da ureia.Contexto: Fluxograma
Sugestão:
1. Reposicione termos e conectores com arraste de mouse;
2. Para um fluxograma diferente no conteúdo, modifique os termos no código;
2. Para um fluxograma diferente no formato, altere as características de fonte e conectores na constante "annotations".
2 Análise de dados
2.1 Inserção de dados do usuário
Contexto
Sugestão:
1. Experimente alterar os dados inseridos, sobrepondo ou não o gráfico;
2. Experimente alterar a representação de dados em "mode" e "type", para pontos, linhas, pontos+linhas, barras.
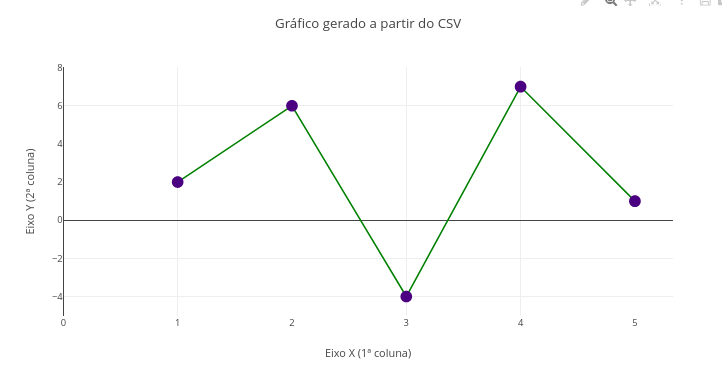
2.2 Carregamento de arquivo para análise
Contexto - Arquivo CSV
Instruções:
- 1 Clique em add plot e selecione um arquivo CSV no botão browse superior que é apresentado. Nota: variável X na 1a. coluna do arquivo, e variável Y na 2a. coluna;
- Clique em add plot novamente para a visualização do gráfico resultante.
Sugestão
1. Experimente outros arquivos CSV;
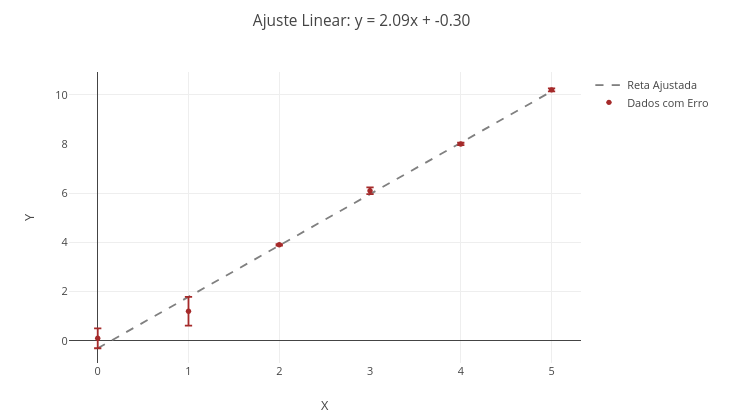
2. Varie os aspectos do gráfico, como tipo, cor, tamanho do marcador, etc.2.3 Ajuste linear de dados
Contexto:
Equação:
\[ y = \alpha x + \beta + \varepsilon \]
Onde:
- y = variável dependente;
- x = variável independente;
- \(\alpha\) = inclinação da reta ajustada (slope);
- \(\beta\) = intercepto da reta ajustada;
- \(\epsilon\) = erro da medida.
\[ \alpha = \frac{n \sum x_i y_i - \sum x_i \sum y_i}{n \sum x_i^2 - \left( \sum x_i \right)^2} \]
\[ \beta = \frac{\sum y_i - \alpha \sum x_i}{n} \]
\[ \hat{y}_i = \alpha x_i + \beta \]
\[ \varepsilon_i = \left| y_i - \hat{y}_i \right| \]
Sugestão:
1. Apresente os pontos sobrepostos ou não à reta do ajuste. Para a sobreposição, escolha "mostrarPontos = true" e "mostrarReta = true";
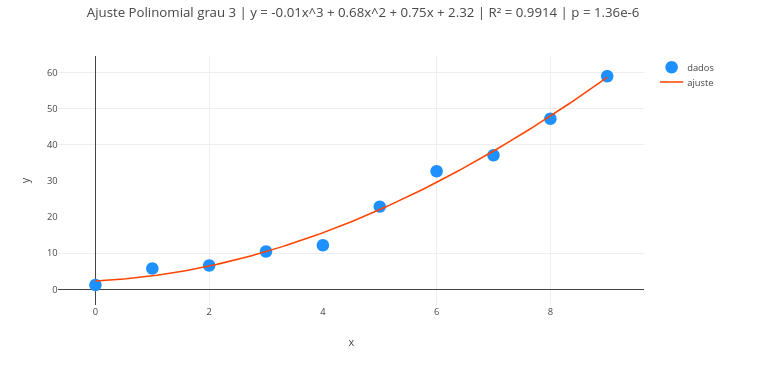
2. Altere os dados e realize novo ajuste, para obtenção de outros parâmetros da reta.2.4 Regressão polinomial
Contexto:
2.4.1 Equação
\[ y = \beta_0 + \beta_1 x + \beta_2 x^2 + \cdots + \beta_g x^g \] | Dessa forma, elabora-se a matriz de Vandermonde como:
\[ X = \begin{bmatrix} 1 & x_1 & x_1^2 & \cdots & x_1^g \\\\ 1 & x_2 & x_2^2 & \cdots & x_2^g \\\\ \vdots & \vdots & \vdots & & \vdots \\\\ 1 & x_n & x_n^2 & \cdots & x_n^g \end{bmatrix} \]
\[
\boldsymbol{\beta} = (X^T X)^{-1} X^T \mathbf{y}
\]
Onde:
- T representa a matriz transposta
Sugestão:
1. Experimente o grau 1 para o polinômio, ou seja, uma redução do tratamento ao ajuste linear;
2. Altere a formatação de rótulos, cores, tamanhos, etc, no código;
3. Sobreponha alguns ajustes, edite e reposicione a legenda;
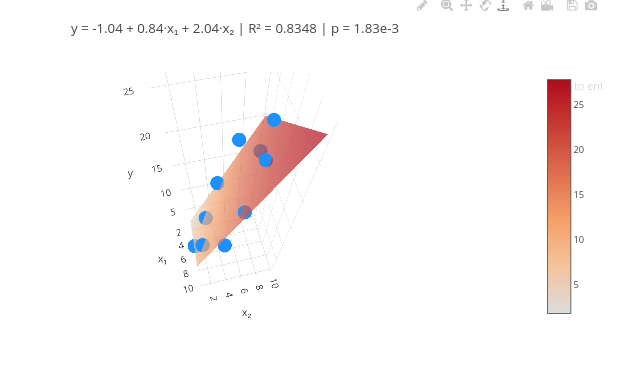
4. Teste o código com outro vetor de dados.2.5 Regressão multilinear
Contexto:
Equação:
\[ y_i = \beta_0 + \beta_1 x_{1i} + \beta_2 x_{2i} + \cdots + \beta_p x_{pi} + \varepsilon_i \]
\[ \mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \boldsymbol{\varepsilon} \]
\[ \hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y} \]
Onde: - \(\beta\) = vetor de coeficientes; - y = vetor de respostas; - X = matriz de projeto; - \(\epsilon\) = ruído aleatório
\[ \hat{\mathbf{y}} = \mathbf{X} \hat{\boldsymbol{\beta}} \] \[ \mathbf{e} = \mathbf{y} - \hat{\mathbf{y}} \]
Instruções
Observer que há um flag booleano (mostrarAjuste) ao início do código: false para dados somente, e true para o ajuste;
Pode-se clicar em add plot para visualizar os dados com o flag em false, seguido de outro add plot com o flag em true.
Sugestão:
1. Resultados de QSAR ("Quantitative Structure-Activity Relationship") utilizam análise por ajuste multilinear para identificar a força das variáveis preditoras. Experimente na internet um conjunto de dados que possua 2 variáveis (ex: concentração, pH, composto A, B, etc.)
2. Para mais variáveis preditoras, veja o código que segue !!2.6 Ajuste linear múltiplo com 3 ou mais variáveis preditoras
Contexto
Sugestão:
1. Experimente variar o número de preditores (xi). Para isso:
a. Sem quiser reduzir, basta retirar o(s) vetor(res) desejados, e corrigir seu quantitativo na linha: "const X = x1.map((_, i)";
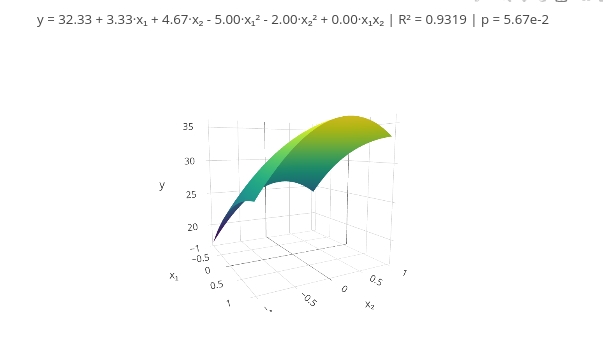
b. Se quiser aumentar, acrescente um novo vetor na linha correspondente, e atualize o quantitativo no mapeamento de "const X = x1.map((_, i)".2.7 Metodologia de Superfície de Resposta (MSR)
Contexto
Equação:
\[ y = \beta_0 + \beta_1 x_1 + \beta_2 x_2 + \beta_{11} x_1^2 + \beta_{22} x_2^2 + \beta_{12} x_1 x_2 + \varepsilon \]
\[ \mathbf{y} = \mathbf{X} \boldsymbol{\beta} + \boldsymbol{\varepsilon} \]
\[
\hat{\boldsymbol{\beta}} = (\mathbf{X}^\top \mathbf{X})^{-1} \mathbf{X}^\top \mathbf{y}
\]
Sugestão:
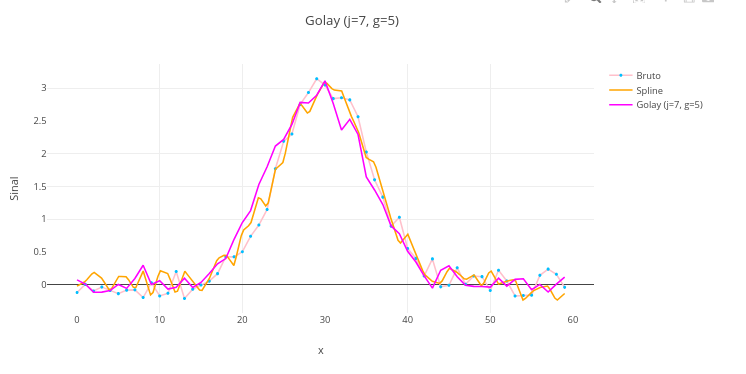
1. Como sugerido anteriormente, experimente utilizar dados da literatura ou de outra fonte cujas respostas já são conhecidas, substituindo os respectivos vetores ao início do código. Isso permite comparar a eficácia de uso da ferramenta apresentada.2.8 Suavização de dados - Spline e filtro de Savitzky-Golay
Contexto - Spline cúbica
Equação
Matematicamente, a spline é aplicada de tal forma que:
\[ S_i(x) = a_i + b_i(x - x_i) + c_i(x - x_i)^2 + d_i(x - x_i)^3 \]
Onde:
- S\(_{i}\)(x) = função spline cúbica no intervalo [xi,xi+1];
- a\(_{i, b\){i}\(, c\){i}\(, d\)_{i}$ = coeficientes específicos para o segmento i;
- x\(_{i}\) = ponto inicial do intervalo;
- x = variável independente.
Contexto - Filtro de Savitzky-Golay
Equação
Matematicamente:
\[ \tilde{y}_i = \sum_{j=-m}^{m} c_j \cdot y_{i+j} \]
Onde:
- y\(_{i}\) = valor suavizado na posição i;
- y\(_{i+j}\) = valores reais da série dentro da janela;
- c\(_{i}\) = coeficientes do filtro derivados de uma regressão polinomial;
- m = número de pontos de cada lado da janela central.
Instruções:
- O código possui dois flags true/false, um para “usarGolay” e outro para “usarSpline”;
- Visualize os dados brutos adotando false para ambos os flags;
- Para sobrepor uma spline cúbica mude sua constante para true;
- Para sobrepor o filtro de Savitzky-Golay, mude sua constante para true;
- Para ajustar o filtro, modifique seus parâmetros nas constantes janela (janela móvel) e/ou grau (grau do polinômio).
Sugestão:
1. Compare os efeitos de suavização pelos dois tratamentos de interpolação polinomial;
2. Aplique apenas o filtro, ajustando parâmetros de janela móvel e grau polinomial;
3. Experimente a interpolação com outros dados brutos no código, substituindo as constantes "x_values" e "y_raw" por vetores numéricos;2.10 Ajuste não linear de dados
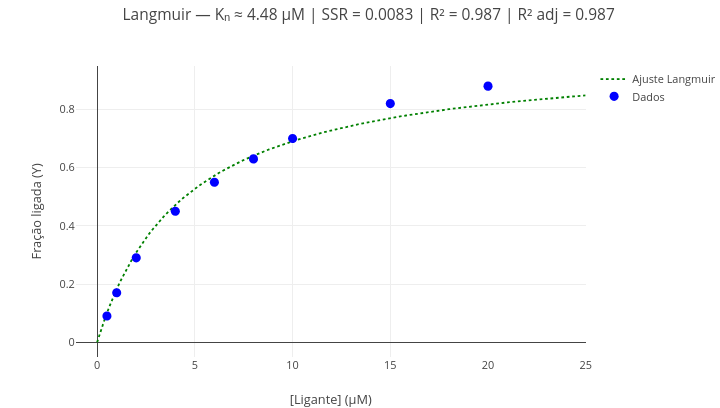
Contexto: Interação ligante-proteína (algoritmo de grid search)
Equação:
\[ Y = \frac{aX}{K_d + X} \] Onde:
- Y = fração de sítios ocupados;
- X = concentração do ligante livre;
- a = valor de saturação (sítios de mesma afinidade completamente ocupados);
- Kd = constante de equilíbrio de dissociação do complexo.
\[ SSR(a, K_d) = \sum_{i=1}^n \left( y_i - \frac{a x_i}{K_d + x_i} \right)^2 \]
Onde:
SSR = soma dos quadrados dos resíduos (inversamente propocional à qualidade do ajuste)
Sugestão:
1. Como no exemplo anterior, apresente alternativamente os pontos e a curva de ajuste pela operação "booleana" ("mostrarPontos = true/false"; "mostrarCurva = true/false");
2. Altere os dados e realize novo ajuste, para obtenção de outros parâmetros da equação (Kd, SSR, R², R² ajustado). Obs: R² adj corresponde ao valor de R² corrigido para o número de parâmetros do modelo (no caso, "2"), incidindo diretamente nos "graus de liberdade" para o ajuste.
Contexto - Inibição competitiva em cinética enzimática (algoritmo de Gauss-Newton)
Equação:
\[ \theta_{k+1} = \theta_k + \left(J^T J\right)^{-1} J^T \cdot r \]
Instruções
- Perceba que o código possui um flag para true/false na constante ajuste. Deixe-o em false para a representação dos dados, somente (vetores x e y, ou S e v);
- Na área da Plot do código, acerte a constante trace para que fique com os vetores de dados, também;
- Clique em add plot para visualizar os pontos experimentais;
- Para superimpor a curva ajustada, atualize as constantes ajuste e trace (vetores S_fit e v_fit), e escolha lines em mode.
3 Aplicativos
3.1 CarboId - Experimentação simulada para caracterização de aminoácidos e cadeias
Contexto:
Instruções do simulador
Opção A: descobrir a natureza de uma amostra:
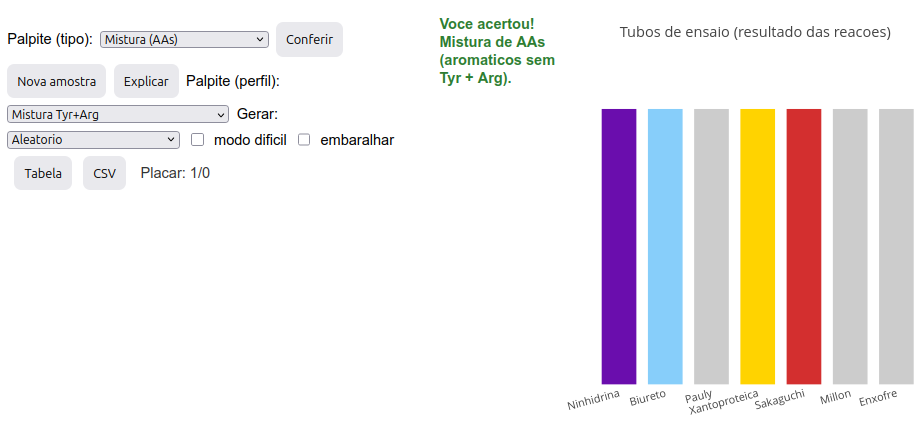
Clique na imagem abaixo e dê um “add”. Será gerado um conjunto de 7 barras coloridas, representando as cores obtidas em tubos de ensaio para 7 reações distintas para caracterização proteica: ninhidrina, biureto, Pauly, xantoproteica, Sakagushi, Millon, e enxofre (Cys);
Descubra o tipo de amostra em função do padrão de cores dos tubos apresentado, fornecendo um palpite. O palpite deve ser por tipo (no menu “Palpite (tipo)” - AA, cadeia, ou mistura de AAs) e por perfil (no menu “Palpite (perfil)” - aleatório, AA(s) específico(s) ou mistura específica);
Após a seleção nos 2 menus, clique em “Conferir”. Se tiver acertado, um “Placar” logo abaixo irá computar o número de acertos/número de amostras “jogadas”;
Se errar, tente de novo ou busque a explicação do padrão de cores em “Explicar”;
Existem duas outras possibilidades: “Modo difícil”, que esconde o nome das reações, embora mantendo sua sequência, e “Embaralhar”, que altera a ordem dos tubos;
Para uma nova amostra, basta clicar em “Nova amostra”;
Opção B: descobrir o padrão de cores de uma reação para uma amostra selecionada:
- Selecione uma amostra em “Gerar”, e clique em “Nova amostra”. Será gerado um conjunto de tubos com o padrão de reações esperado para a amostra.
Sugestão:
1. Experimente clicar em "Nova amostra" para verificar a frequência de possibilidades;
2. Experimente as duas opções de operação do "jogo" (sim, é um jogo...afinal, tem um placar!);
3. Experimente o "modo difícil" (sem referência às reações em sequência) ou "embaralhar" (permuta da ordem das reações). Por óbvio, contudo, evite os dois simultaneamente !
3.2 ZeMol - Visualizador 3D para modelos moleculares
Contexto:
- modelos carregados online do site PDB ou PubChem;
- modelos carregados da memória física do dispositivo (formatos PDB, MOL, SDF, XYZ, TXT);
- informações de identificação do arquivo PDB (ou nome do arquivo PubChem);
- cabeçalho contendo quantitativo de cadeias, ligantes, íons, e ligações dissulfeto;
- identificação apenas dos carbonos \(\alpha\) de cada resíduo do esqueleto polipetídico, facilitando a visualização do modelo;
- visualização automática de ligações dissulfeto;
- identificação de resíduos, ligantes, e íons, por hover de mouse sobre o modelo;
- checkbox para visualização de estrutura secundária de proteínas (hélice, folha-\(\beta\));
- checkbox para índice de polaridade dos resíduos;
- sliders para tamanho do carbono CA (PDB) ou átomos no geral, e opacidade do modelo (útil pra destaque de fendas, estrutura 2a., cadeia carbônica, ligantes, íons);
- ocultar/mostrar partes distintas do modelo, clicando-se em sua legenda (cadeia, íons, hélice/folhas, ligantes);
- Destaque de AAs (individual, sequência, todos de um tipo, grupos pré-definidos no script);
- HTML autosuficiente no salvamento, “congelando” o modelo nas características que se deseja apresentar;
- Zoom em dispositivos móveis (um dedo fixo na tela e outro arrastando o modelo) e Pan (deslocamento do modelo com 2 dedos na tela) - ações também presentes nos ícones acima do modelo;
- ajustes facultativos no próprio script (tamanhos, cores, espessuras, grupos de AAs, tipo e espessura de traços, por ex).
Sugestão:
1. Carregue um modelo com código PDB, ou uma molécula do site PubChem;
2. Se proteína, experimente os efeitos separados e combinados dos "sliders" para tamanho do CA e opacidade;
3. Observe a polaridade dos resíduos proteicos no "checkbox" homônimo;
4. Experimente ampliar/reduzir ou deslocá-lo na tela com auxílio do mouse, ou fazê-lo num smartphone conforme a instrução dada acima;
5. Mostre ou esconda alternativamente cada cadeia ou todas, ligantes e íons, clicando-se nos termos respectivos da legenda;
6. Selecione resíduos de interesse do modelo, como sítio catalítico, de ligação a coenzimas, ou de regulação da atividade. Para isso, digite no campo correlato e clique em "destacar". Algumas sugestões:
a. Individuais: Arg15, ASP117, 15, A:15, A:ARG15;
b. Faixas: 15-30, A:15-30, Arg15-Asp30;
c. Múltiplos separados por vírgula: "Arg15, Asp117";
d. Grupos: "aromatico", "polar", "apolar", "basico", "acido", "small".
3.3 ProtId - Experimentação simulada para caracterização de carboidratos
Contexto:
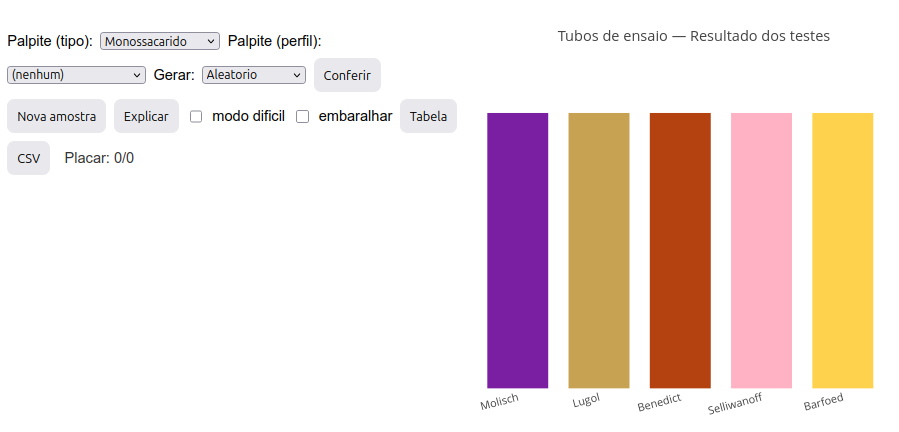
Teste de Molish - detecta carboidratos (anel púrpura); reação com \(\alpha\)-naphtol produzindo furfural ou hidroximetilfurfural;
Teste de lugol (I\(_{2}\)/KI) - detecta polissacarídios (tubo azul escuro);
Teste de Benedict - detecta monossacarídos redutores (e também lactose); tubo com precipitado laranja escuro/tijolo;
Teste de Selliwanoff (resorcinol) - diferencia cetoses (tubo vermelho) de aldoses (tubo rosa fraco);
Teste de Barfoed - diferencia mono (tubo com precipitado vermelho - óxido cuproso) de oligossacarídios (tubo amarelado).
Instruções do simulador
Opção A: descobrir a natureza de uma amostra:
Clique na imagem abaixo e dê um “add”. Será gerado um conjunto de 5 barras coloridas, representando as cores obtidas em tubos de ensaio para as 5 reações distintas para caracterização de carboidratos: Molish, Lugol, Benedict, Selliwanoff, e Barfoed.
Descubra o tipo de amostra em função do padrão de cores dos tubos apresentado, fornecendo um palpite. O palpite deve ser por tipo (no menu “Palpite (tipo)” - monossacarídio, di-oligossacarídio, polissacarídio, água. E por perfil (no menu “Palpite (perfil)” - redutor, não redutor, aldose, cetose, monossacarídio, di-oligossacarídio, e polissacarídio;
Após a seleção nos 2 menus, clique em “Conferir”. Se tiver acertado, um “Placar” logo abaixo irá computar o número de acertos/número de amostras “jogadas”;
Se errar, tente de novo ou busque a explicação do padrão de cores em “Explicar”;
Existem duas outras possibilidades: “Modo difícil”, que esconde o nome das reações, embora mantendo sua sequência, e “Embaralhar”, que altera a ordem dos tubos;
Para uma nova amostra, basta clicar em “Nova amostra”;
Opção B: descobrir o padrão de cores de uma reação para uma amostra selecionada:
- Selecione uma amostra em “Gerar”, e clique em “Nova amostra”. Será gerado um conjunto de tubos com o padrão de reações esperado para a amostra.
Sugestão:
1. Experimente clicar em "Nova amostra" para verificar a frequência de possibilidades;
2. Experimente as duas opções de operação do "simulador/aplicativo/jogo");
3. Experimente o "modo difícil" (sem referência às reações em sequência) ou "embaralhar" (permuta da ordem das reações). Por óbvio, contudo, evite os dois simultaneamente !
3.4 LeadHunt - Prospecção para composto líder como inibidor enzimático
Observação: o LeadHunt possui códigos internos para estilos e montagens do painel, e que interferem no bootstratp do criador de páginas web utilizado para o Bioquanti (pacote Quarto para R). Nesse sentido, clique na imagem abaixo para baixá-lo, e depois abrar-o num navegador.
Instruções gerais
- No campo superior do app há 4 botões:
- reset - renova a simulação para outros dados e gráficos;
- html - salva os gráficos e dados em arquivo interativo;
- diagnóstico - abre janela popup para o diagnóstico do modelo de inibição e valores de parâmetros;
- parâmetros - abre janela para simulação gráfica frente a parâmetros do usuário.
- Há dois modos para estudo para uso do LeadHunt: inserindo-se parâmetros cinéticos e termodinâmicos para observar os resultados expressos em gráficos (botão parâmetros), ou caracterizar os parâmetros a partir dos gráficos (botão diagnóstico).
Parâmetros
- Ki, constante de equilíbrio de dissociação do inibidor (desejável < 10\(\mu\)M);
- Vmax, velocidade limite da reação enzimática;
- Km, constante de Michaelis-Mentem;
- \(\Delta\)H, variação de entalpia de ligação com inibidor;
- \(\Delta\)S, variação de entropia de ligação com inibidor;
- \(\Delta\)Cp, variação de capacidade calorífica da ligação com inibidor;
- MW, peso molecular;
- HAC, número de átomos pesados (todos, exceto hidrogênio);
- HBD, número de doadores de ligação de H;
- HBA, número de aceptores de ligação de H;
- logP, logaritmo do coeficiente de partição da molécula
- LE, eficiência de ligante (ligand efficiency):
\[ LE = \frac{\Delta G}{HAC}; \]
\[
LE = 1.37 \frac{pIC_{50}}{HAC}
\]
Onde pIC\(_{50}\) = -log(IC\(_{50}\));
- LLE, eficiência lipofílica do ligante (lipophilic ligand efficiency; valores >5 são considerados bons),
\[ LLE=pIC_{50}-logP \]
- BEI, índice de eficiência da ligação (binding efficiency index),
\[
BEI=\frac{pIC_{50}}{MW}
\]
Onde: MW, kDa; faixas típicas pela regra de Lipinski (regra dos 5): MW<=500, HBD<=5, HBA<=10, cLogP<=5. E também LE >= 0.3; LLE >= 5.
- IC\(_{50}\), concentração do inibidor que confere 50% de resposta (adequado quando < 10\(\mu\)M);
- nH, constante de Hill (índice de cooperatividade)
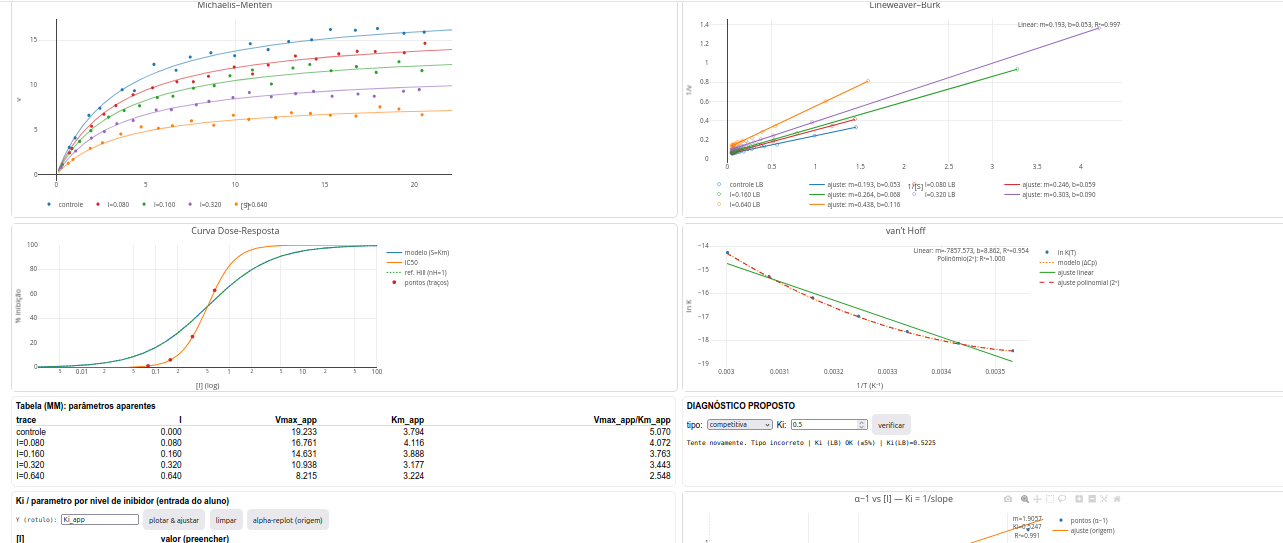
Uso do LeadHunt para identificação de um composto líder
O tipo de inibição reversível (competitiva, incompetitiva, não competitiva pura, não competitiva mista);
O valor de Ki por construção de gráficos secundários variados a partir da tabela de preenchimento de parâmetros (Tabela para Ki) - plota-se a concentração I do inibidor versus Km, Vm, ou Km/Vm aparentes. Alternativamente, plota-se I contra o valor de (\(\alpha\)-1), onde \(\alpha\) representa “1+I/Ki”;
O valor de IC\(_{50}\) e de nH (inclinação do ponto médio) pela curva de dose-resposta do inibidor (estimativa de cooperatividade na ligação);
O valor de \(\Delta\)H e \(\Delta\)S para o plot de Van’t Hoff. No caso em que o gráfico apresentar-se curvilinear, também o valor valor de \(\Delta\)Cp;
Os valores de LE e LLE;
Avaliar o candidato junto às regras de Lipinski.
Sugestão
1. Compare os tipos de inibição enzimática por inspeção visual dos gráficos de Michaelis-Mentem e de Lineweaver-Burk, dando ênfase às variações ou não de "Km" e "Vmax" entre esses;
2. Calcule o valor de "Ki" por métodos distintos utilizando-se o campo de preenchimento para "replot" por nível de inibidor (tabela de "Parâmetros aparentes"):
a) obtendo-se o valor de "1/Ki" para plot de I x (alfa-1) do botão "alfa-replot" (inclinação da reta);
b) por intercepto "-Ki" a partir de plots de I x Vm_app, Km_app, ou Km/Vm_app (dependendo do modelo de inibição);
c) por "inclinação" ou "intercepto" de replots de Lineweaver-Burk contra I
3. Experimente o modo "parâmetros" com valor de "nH" diferente de 1 (ex: 2,8), para avaliar o comportamento da curva dose-reposta sob efeitos de cooperatividade;
4. Experimente o modo "parâmetros" com valor para capacidade calorífica diferente de zero (ex: -2000), para avaliar a possibilidade de efeitos secundários na ligação em função da temperatura, como transição conformacional da enzima;
5. Teste a curvatura do plot de Van´t Hoff para variação de capacidade calorífica da interação, fornecendo valores positivos (ex: 2000) e negativos (ex: -2000): se concavidade para cima, valor negativo da variação de capacidade calorífica; se concavidade para baixo, valor positivo.Referências:
- Marangoni, Alejandro G. Enzyme kinetics: a modern approach. John Wiley & Sons, 2003.
- Copeland, Robert A. Evaluation of enzyme inhibitors in drug discovery: a guide for medicinal chemists and pharmacologists. John Wiley & Sons, 2013.
- Copeland, Robert A. Enzymes: a practical introduction to structure, mechanism, and data analysis. John Wiley & Sons, 2023.
- Leone, Francisco A. Fundamentos de cinética enzimática. Editora Appris, 2021.
3.5 Gizz - Lousa Digital
- Pode ser exportada como arquivo autossuficiente HTML em botão da própria lousa, e permitindo compartilhamento da imagem gerada, bem como continuidade de sua edição;
- Faculta a personalização do código que o produz, permitindo inserir/alterar canetas, espessuras, e ações distintas não previstas no código-fonte.
- Escrever com 7 cores e 3 espessuras distintas;
- Acessar 4 planos de fundo de coloração diferente;
- Sobrepor o plano com um grid;
- Utilizar um apagador (erase);
- Utilizar um apontador laser para localização transiente do que se pretende assinalar;
- Salvar a imagem como PNG, indicando data e hora;
- Comandos para desfazer (undo) e de limpeza (clean) do quadro;
- Atalhos de teclado: E=borracha; L=laser; Z desfaz, X limpa, H salva HTML
Sugestão:
1. Pode-se alterar a espessura das canetas em "var baseWidth = 1;" ;
2. Os botões 1x, 2x, 4x são multiplicadores; edite as linhas de criação dos botões pra trocar rótulo/multiplicador (ex.: "addSize('3x', 3, true)").