# Inibição por excesso de S
S <- seq(0, 10, 0.1)
v_alos <- function(S, Vm = 10, Km = 0.5, Ks = 2) {
Vm * S / (S * (1 + S / Ks) + Km)
}
curve(v_alos, xlim = c(0, 10), xlab = "S", ylab = "v")
\[ v=\frac{Vm*S}{S(1+\frac{S}{Ks})+Km} \tag{1}\]
# Inibição por excesso de S
S <- seq(0, 10, 0.1)
v_alos <- function(S, Vm = 10, Km = 0.5, Ks = 2) {
Vm * S / (S * (1 + S / Ks) + Km)
}
curve(v_alos, xlim = c(0, 10), xlab = "S", ylab = "v")
\[ v=\frac{Vm*S}{Km(1+\frac{I}{Kic})+S(1+\frac{I}{Kiu})} \tag{2}\]
R as cuvas michaelianas para modelos clássicos de inibição, considerando valores para as constantes de equilíbrio de dissociação dos inibidores como Kic = 0.2, e Kiu = 1, como no trecho de código abaixo.# Inibição clássica & Michaelis-Mentem
par(mfrow = c(2, 2)) # divide a área de plotagem
S <- seq(0, 10, 0.1) # geração de teores de S
contr <- function(S, Vm = 10, Km = 0.5) {
Vm * S / (Km + S)
} # função de MM, sem inibição
curve(contr, xlim = c(0, 10), xlab = "S", ylab = "v", main = "Competitiva")
# cuva controle; veja que o título tem que ser adicionado para o 1a. de par
# de curvas, controle e inibição
# Modelos de inibição:
# Competitiva
comp.i <- function(S, Vm = 10, Km = 0.5, I = 2, Kic = 0.2) {
Vm * S / (Km * (1 + I / Kic) + S)
}
curve(comp.i, add = TRUE, col = "red", lty = 2) # competitiva
# Não competitiva pura
pura.i <- function(S, Vm = 10, Km = 0.5, I = 2, Ki = 1) {
Vm * S / (Km * (1 + I / Ki) + S * (1 + I / Ki))
}
curve(contr, xlim = c(0, 10), xlab = "S", ylab = "v", main = "Não Compet. Pura")
curve(pura.i, add = TRUE, col = "red", lty = 2) # não competitiva pura (Kiu=Kic=Ki)
# Não competitiva mista
mista.i <- function(S, Vm = 10, Km = 0.5, I = 2, Kic = 0.2, Kiu = 1) {
Vm * S / (Km * (1 + I / Kic) + S * (1 + I / Kiu))
}
curve(contr, xlim = c(0, 10), xlab = "S", ylab = "v", main = "Não Compet. Mista")
curve(mista.i, add = TRUE, col = "red", lty = 2) # não competitiva mista
# Incompetitiva
incomp.i <- function(S, Vm = 10, Km = 0.5, I = 2, Kiu = 1) {
Vm * S / (Km + S * (1 + I / Kiu))
}
curve(contr, xlim = c(0, 10), xlab = "S", ylab = "v", main = "Incompetitiva")
curve(incomp.i, add = TRUE, col = "red", lty = 2) # incompetitiva
layout(1) # retorna à janela gráfica original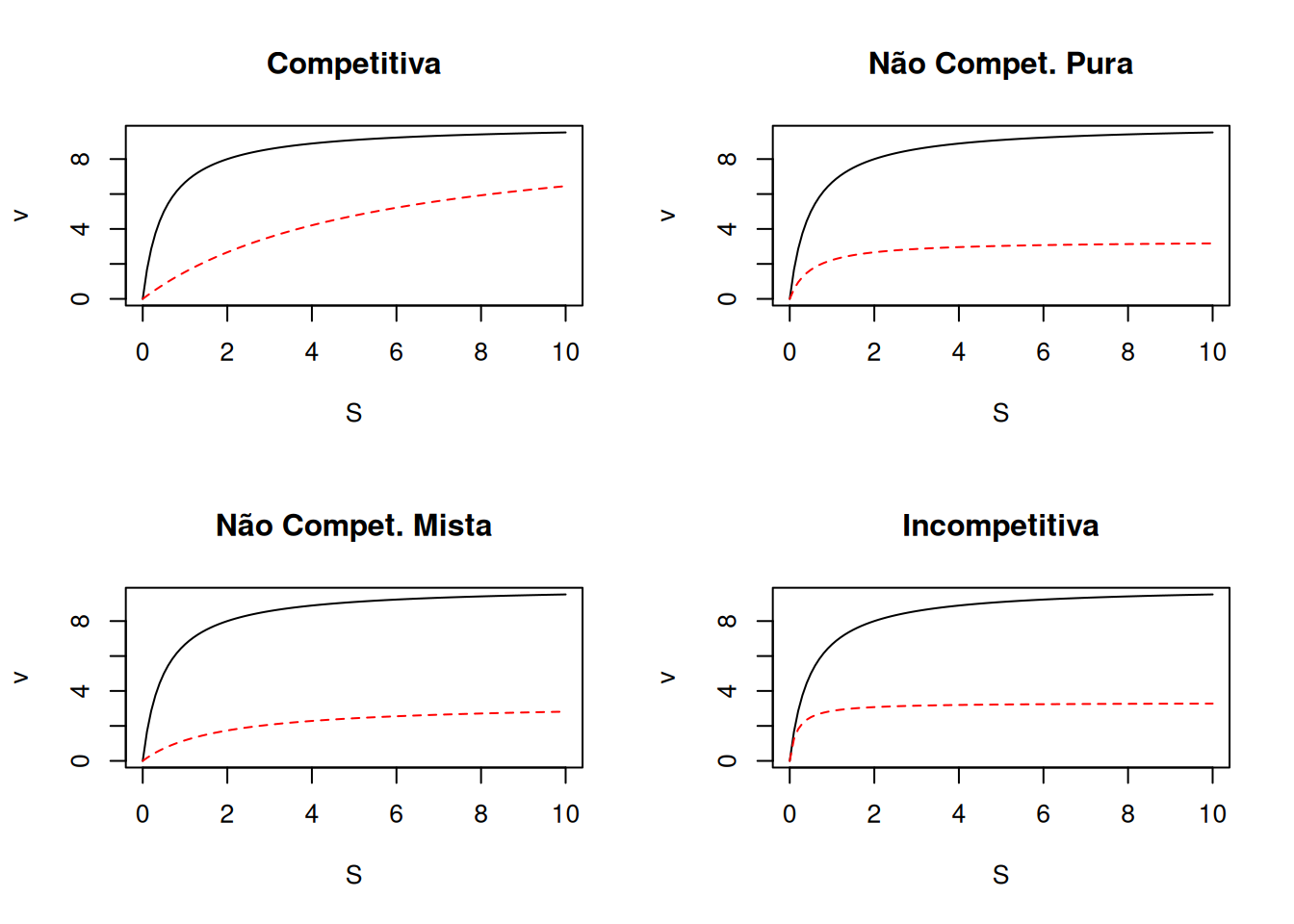
\[ \frac{1}{v}=\frac{1}{Vm}+\frac{Km(1+\frac{I}{Kic})}{Vm}*\frac{1}{S} \quad ;\, competitivo \tag{3}\]
\[ \frac{1}{v}=\frac{1}{Vm}+\frac{Km(1+\frac{I}{Ki})}{Vm}*\frac{1}{S(1+\frac{I}{Ki})} \quad ;\, puro \tag{4}\]
\[ \frac{1}{v}=\frac{1}{Vm}+\frac{Km(1+\frac{I}{Kic})}{Vm}*\frac{1}{S(1+\frac{I}{Kiu})} \quad ;\, misto \tag{5}\]
\[ \frac{1}{v}=\frac{1}{Vm}+\frac{Km}{Vm}*\frac{1}{S(1+\frac{I}{Kiu})} \quad ;\, incompetitivo \tag{6}\]
R junto à transformação de Lineweaver-Burk (ou qualquer outra), como abaixo.# Diagnóstico de inibição por Lineweaver-Burk
# Substrato e Inibidor
S <- seq(0.1, 10, length = 10) # cria um vetor para substrato
I <- 2 # concentração de inibidor
# Parâmetros cinéticos:
Km <- 0.5
Vm <- 10
Kic <- 0.2
Ki <- 0.2
Kiu <- 1
# Equações
v <- Vm * S / (Km + S) # equação de MM
v.comp <- Vm * S / (Km * (1 + I / Kic) + S) # competitivo
v.puro <- Vm * S / (Km * (1 + I / Ki) + S * (1 + I / Ki))
# não competitivo puro
v.misto <- Vm * S / (Km * (1 + I / Kic) + S * (1 + I / Kiu))
# não competitivo misto
v.incomp <- Vm * S / (Km + S * (1 + I / Kiu))
# Gráficos
par(mfrow = c(2, 2)) # área de plot pra 4 gráficos
plot(1 / S, 1 / v, type = "l", main = "Competitivo", ylim = c(0, 2))
points(1 / S, 1 / v.comp, type = "l", col = "red")
plot(1 / S, 1 / v, type = "l", main = "Puro", ylim = c(0, 5))
points(1 / S, 1 / v.puro, type = "l", col = "red")
plot(1 / S, 1 / v, type = "l", main = "Misto", ylim = c(0, 2))
points(1 / S, 1 / v.misto, type = "l", col = "red")
plot(1 / S, 1 / v, type = "l", main = "Incompetitivo", ylim = c(0, 1))
points(1 / S, 1 / v.incomp, type = "l", col = "red")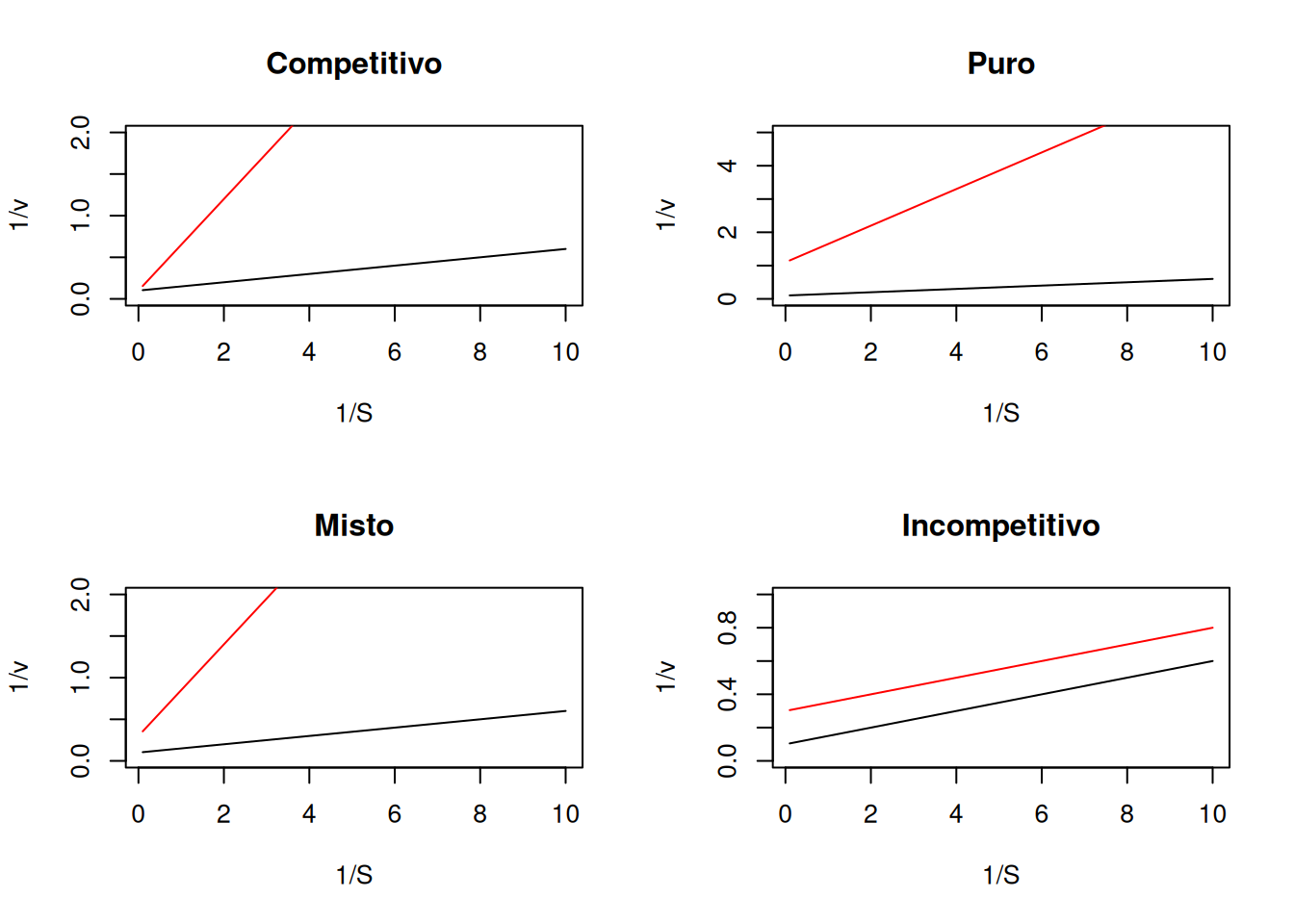
\[ IC_{50} = \frac{(1+\frac{S}{Km})}{(\frac{1}{Kic})+(\frac{1}{Km*Kiu})} \tag{7}\]
\[ IC_{50} = Kic(1+\frac{S}{Km}) \tag{8}\]
R a obtenção de IC\(_{50}\), utilizando-se um ajuste não linear para a equação de quatro parâmetros que segue (curva de Rodbard, DeLean, Munson, e Rodbard (1978)).\[ ativ. residual \, \% =\frac{v}{Vm} = inf+\frac{sup-inf}{1+log(\frac{I}{IC_{50}})^{nH}}) \] {#eq:eqRodb}
# Ajuste não-linear para curva de IC50
logI.nM <- c(5.5, 5.2, 4.9, 4.6, 4.3, 3.7, 3.3, 3, 2.8)
# conc. de I, em unidade log10
ativ.res <- c(0.02, 0.07, 0.12, 0.22, 0.36, 0.53, 0.67, 0.83, 0.85)
# ativ. residual, v/Vm
dados <- data.frame(logI.nM, ativ.res) # criação do dataframe
plot(ativ.res ~ logI.nM, dados) # plot dos dados
ic50.fit <- nls(formula(ativ.res ~ inf + (sup - inf) /
(1 + (logI.nM / logIC50)^nH)),
algorithm = "port", data = dados,
start = list(inf = 0, sup = 0.80, logIC50 = 4, nH = 10),
lower = c(inf = -Inf, sup = -Inf,
logIC50 = 0, nH = -Inf)) # ajuste não linear
summary(ic50.fit) # sumário do ajuste
Formula: ativ.res ~ inf + (sup - inf)/(1 + (logI.nM/logIC50)^nH)
Parameters:
Estimate Std. Error t value Pr(>|t|)
inf -0.3211 0.2932 -1.095 0.32348
sup 1.1200 0.2311 4.847 0.00469 **
logIC50 4.0807 0.2309 17.675 1.06e-05 ***
nH 4.0540 1.7462 2.322 0.06792 .
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.02769 on 5 degrees of freedom
Algorithm "port", convergence message: relative convergence (4)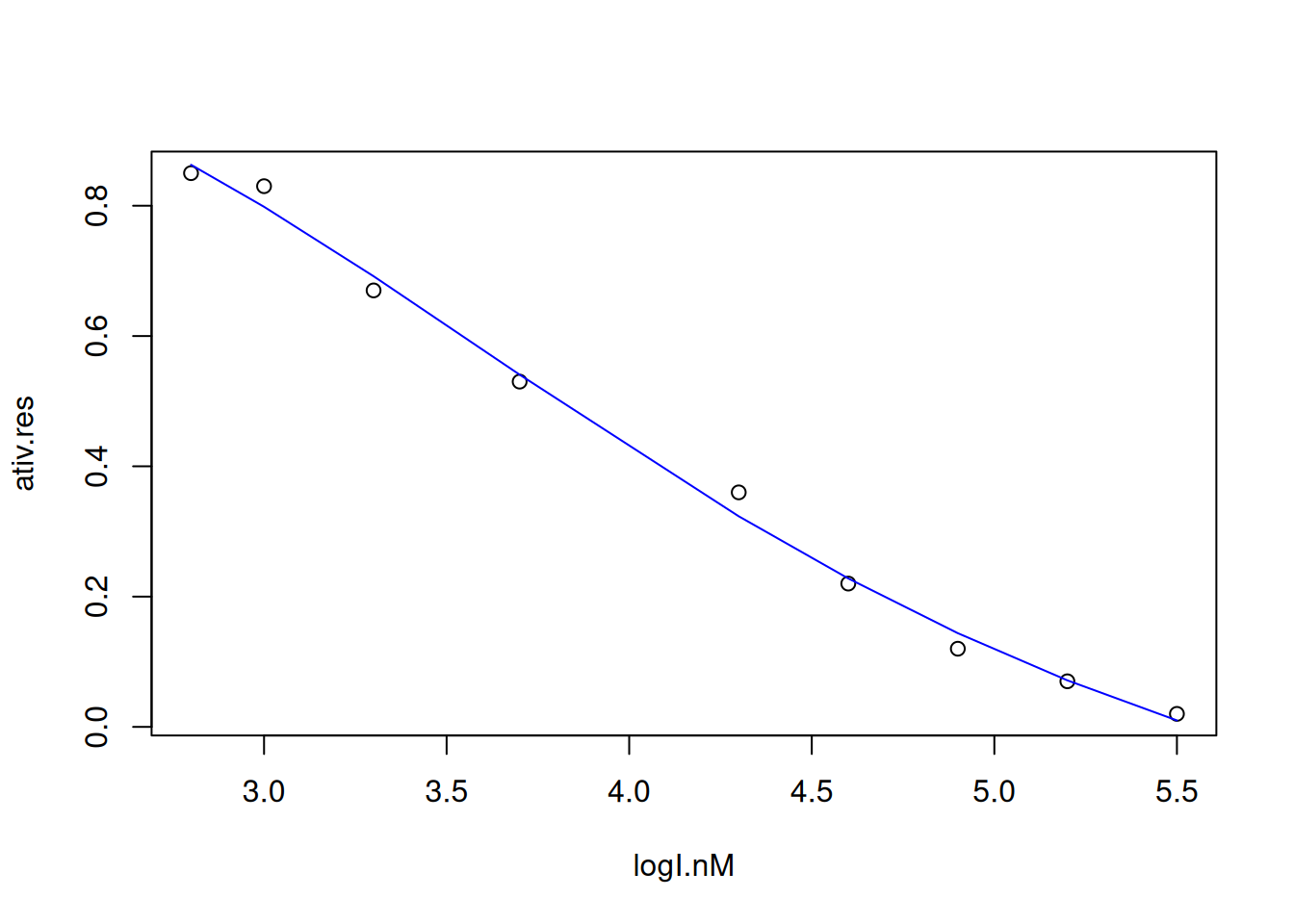
# E para extrair o valor de IC50...
IC50 <- 10^(coef(ic50.fit)[3]) # extração do 3o. parâmetro da tabela
# de ajuste, isto é: logIC50:
IC50 logIC50
12042.04 coef. Isto é muito útil quando desejamos utilizar um coeficiente obtido em cálculos automáticos (programáveis), como veremos mais adiante. Por ora, faz-se interessante apresentar o parâmetro de IC50 obtido de forma mais elegante.R para exprimir resultados quantitativos junto à caracteres (palavras, frases): print() e cat. O trecho de código abaixo ilustra esse output, e algumas diferenças.Valor de IC50 (nM): 12042.04 [1] "Valor de IC50 (nM): 12042.0403466162"print exibe aspas e indexa o nome da coluna, enquanto cat os omite. Em adição, pode-se perceber outra variação no formato de impressão entre os dois comandos pelo exemplo abaixo:[1] "teores: 10" "teores: 25" "teores: 50"teores: 10 25 50R é a de se reduzir o número de casas decimais apresentados. Nesse caso, pode-se utilizar o comando round.[1] "Valor de IC50 (nM): 12042.04"R para o cálculo de IC50, entre esse o pacote drc (dose-response curve).BIC ou da função AIC do R, e que respectivamente calculam valores para o Critério de Informação Bayseiano (Spiess e Neumeyer 2010) ou do Critério de Informação de Akaike (Akaike 1974). Em comum esse parâmetros calculam um valor relativo de informação não computada por um modelo avaliado. O menor valor encontrado para ambos espelha a solução do melhor modelo de ajuste.\[ BIC = p*ln(n)-2*ln(RSE)\\ \\ AIC = n*ln(\frac{RSE}{n})+2k+[\frac{2k(k+1)}{n-k-1}])\\ \tag{9}\]
Onde p representa o no. de parâmetros do modelo, n o número total de pontos experimentais, k o fator p+1, e RSE o valor da soma dos quadrados dos resíduos (residual sum squares).
nlstools, provendo o ajuste, plotagem, inspeção de resíduos, e aplicação de BIC e AIC:# Aplicação de critérios de informação para ajuste de curvas cinéticas
library(nlstools)
comp <- nls(compet_mich, vmkmki, list(Km = 1, Vmax = 20, Ki = 0.5))
# ajuste competitivo, com dados, equação e sementes fornecidas
# pelo pacote nlstools
plotfit(comp, variable = 1) # comando de plotagem do pacote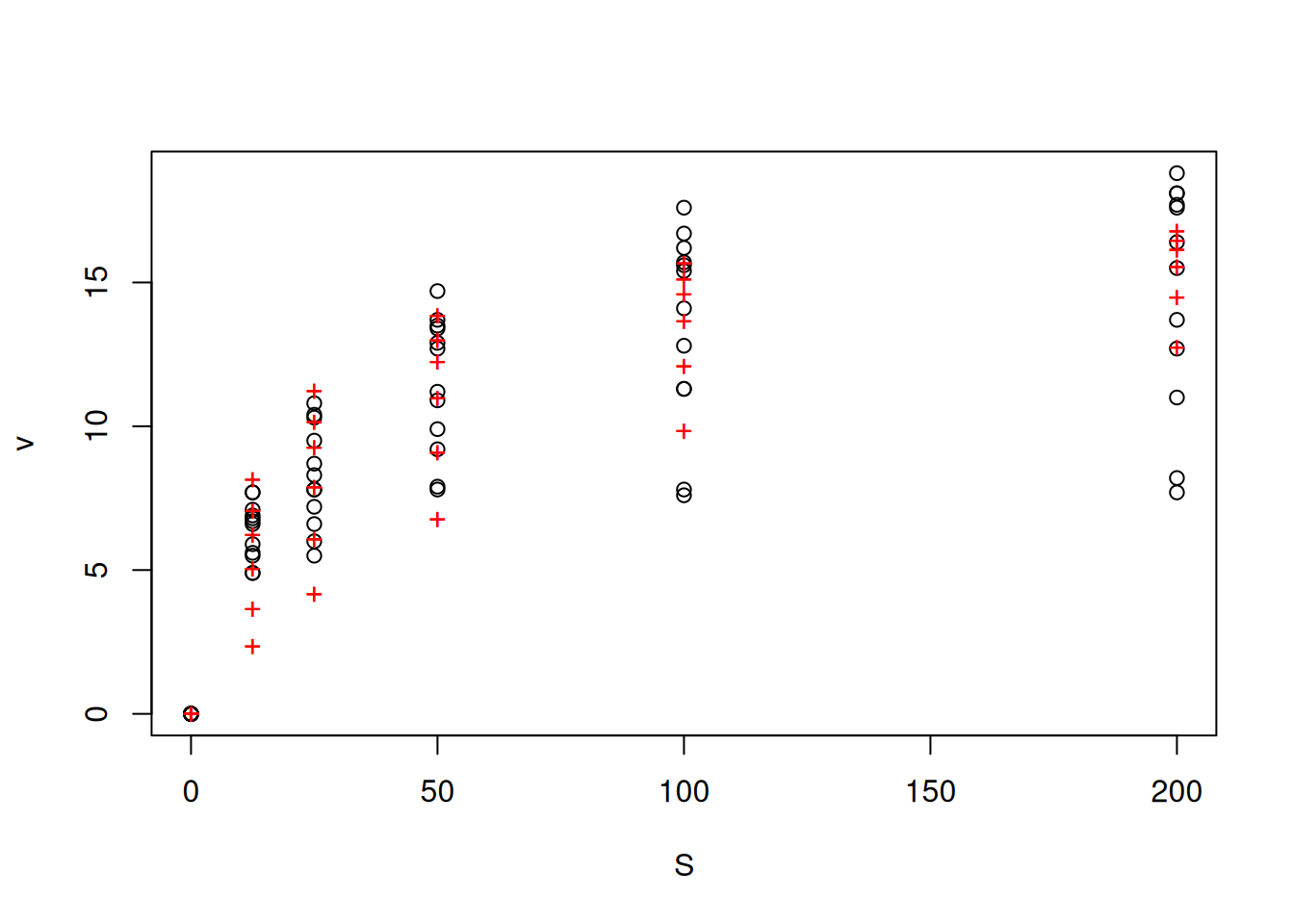
Formula: v ~ S/(S + Km * (1 + I/Ki)) * Vmax
Parameters:
Estimate Std. Error t value Pr(>|t|)
Km 15.2145 2.5005 6.085 5.79e-08 ***
Vmax 18.0557 0.6288 28.713 < 2e-16 ***
Ki 22.2822 4.9060 4.542 2.30e-05 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.603 on 69 degrees of freedom
Number of iterations to convergence: 11
Achieved convergence tolerance: 5.116e-06res_comp <- nlsResiduals(comp) # resíduos do ajuste
plot(res_comp, which = 1) # plotagem de resíduos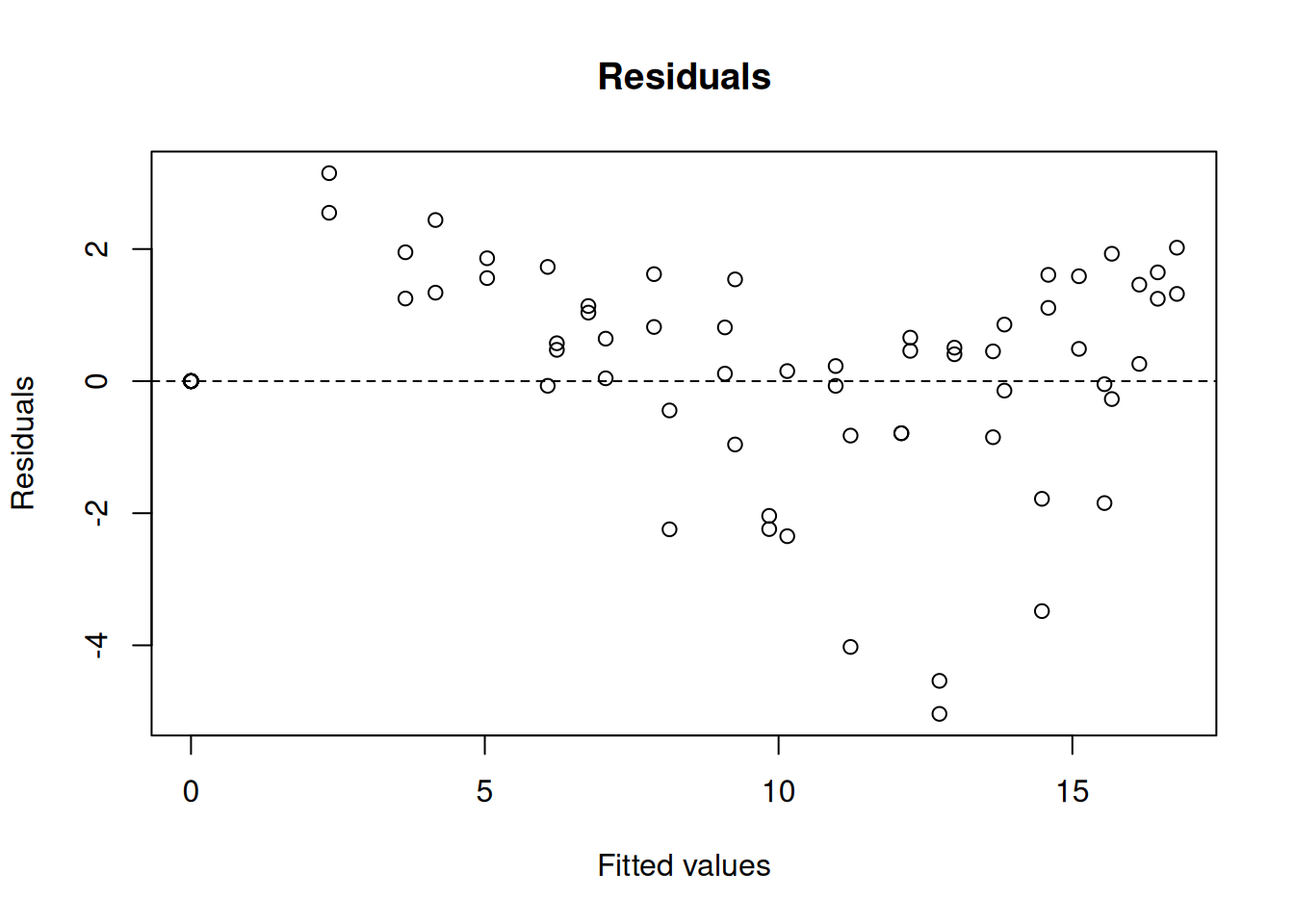
noncomp <- nls(non_compet_mich, vmkmki, list(Km = 1, Vmax = 20, Ki = 0.5))
# o mesmo que acima, mas para o modelo não competitivo
plotfit(noncomp, variable = 1)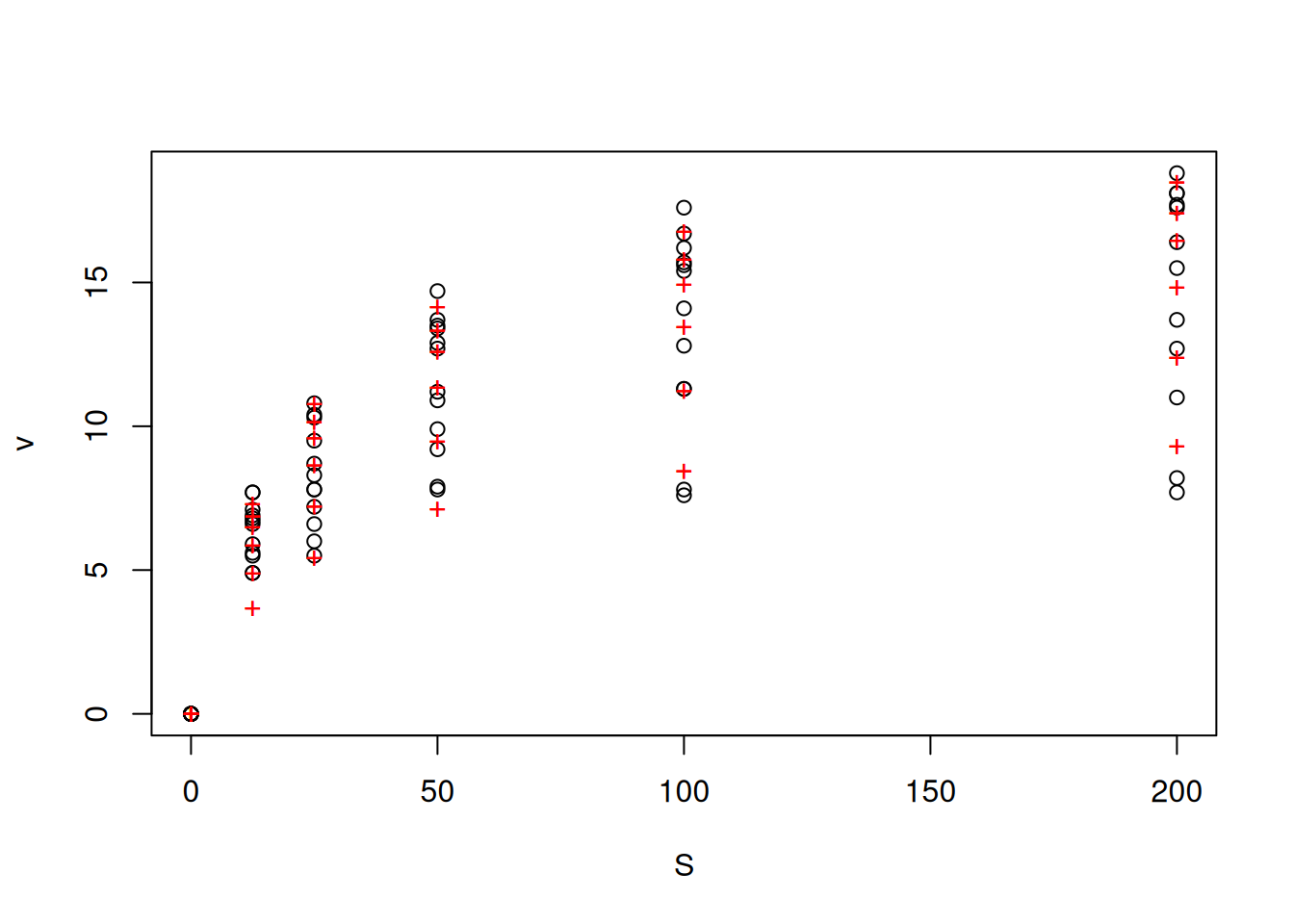
Formula: v ~ S/((S + Km) * (1 + I/Ki)) * Vmax
Parameters:
Estimate Std. Error t value Pr(>|t|)
Km 22.7787 1.4738 15.46 <2e-16 ***
Vmax 20.5867 0.4306 47.80 <2e-16 ***
Ki 101.3563 7.3303 13.83 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.8925 on 69 degrees of freedom
Number of iterations to convergence: 7
Achieved convergence tolerance: 8.27e-06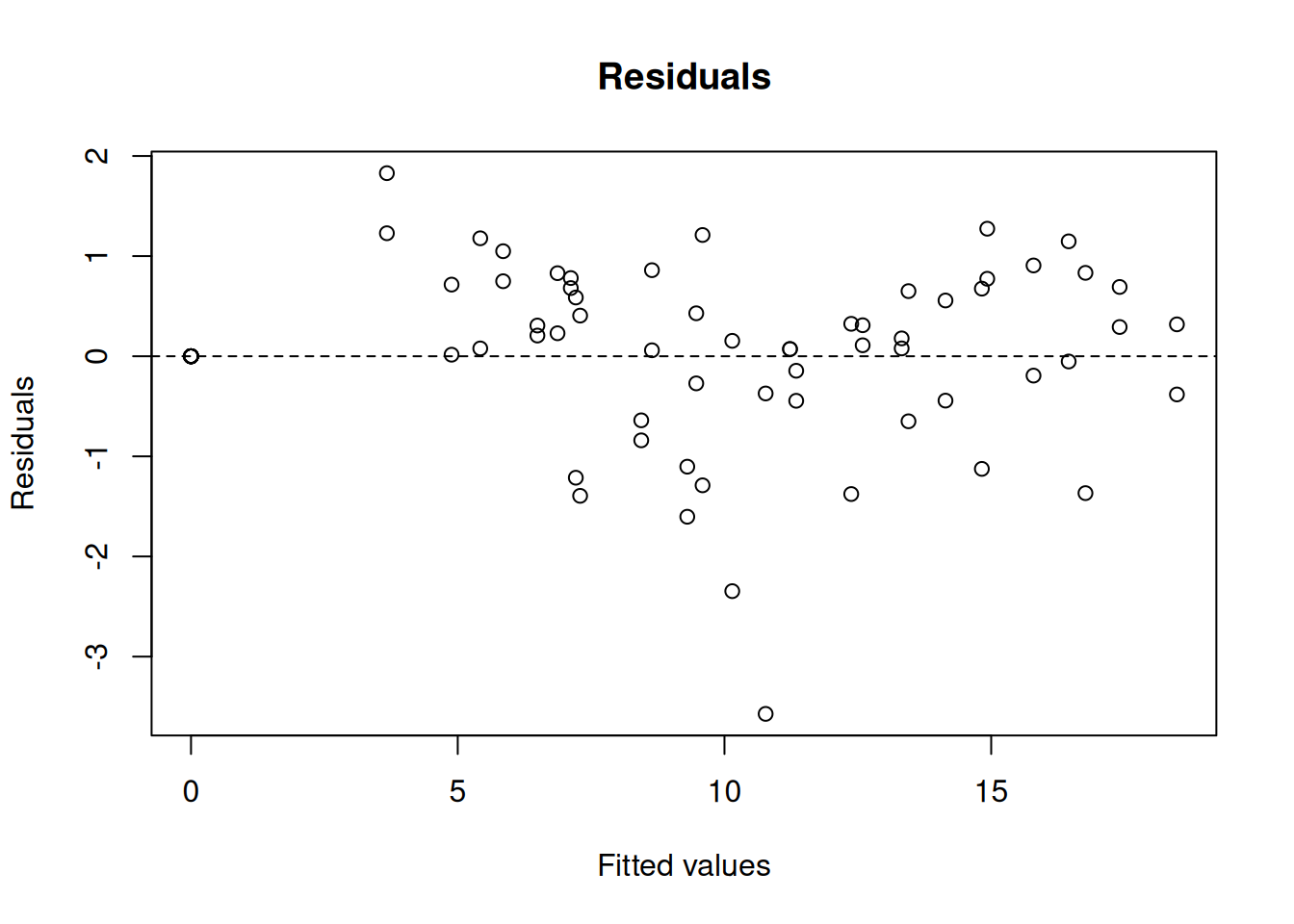
df BIC
comp 4 286.2994
noncomp 4 201.9981 df AIC
comp 4 277.1928
noncomp 4 192.8915\[ E+S \begin{array}{c} _{k1}\\ \rightleftharpoons\\ ^{km1} \end{array} E*S \begin{array}{c} _{k2}\\ \rightleftharpoons\\ ^{km2} \end{array} E*P \begin{array}{c} _{k3}\\ \rightarrow \\ ^{}\end{array} E+P \tag{10}\]
\[ kobs=k2+km2+k3 \tag{11}\]
\[ Ao=\frac{k2*(k2+km2)}{kobs^2} \tag{12}\]
\[ kcat=\frac{k2*k3}{kobs} \tag{13}\]
Onde kobs e Ao representam parâmetros experimentais de constante de velocidade observada e amplitude, respectivamente. Esses parâmetros podem ser obtidos a partir do ajuste não linear da equação abaixo aos dados experimentais:
\[ P=Ao(1-e^ {-kobs} + kcat * t) \tag{14}\]
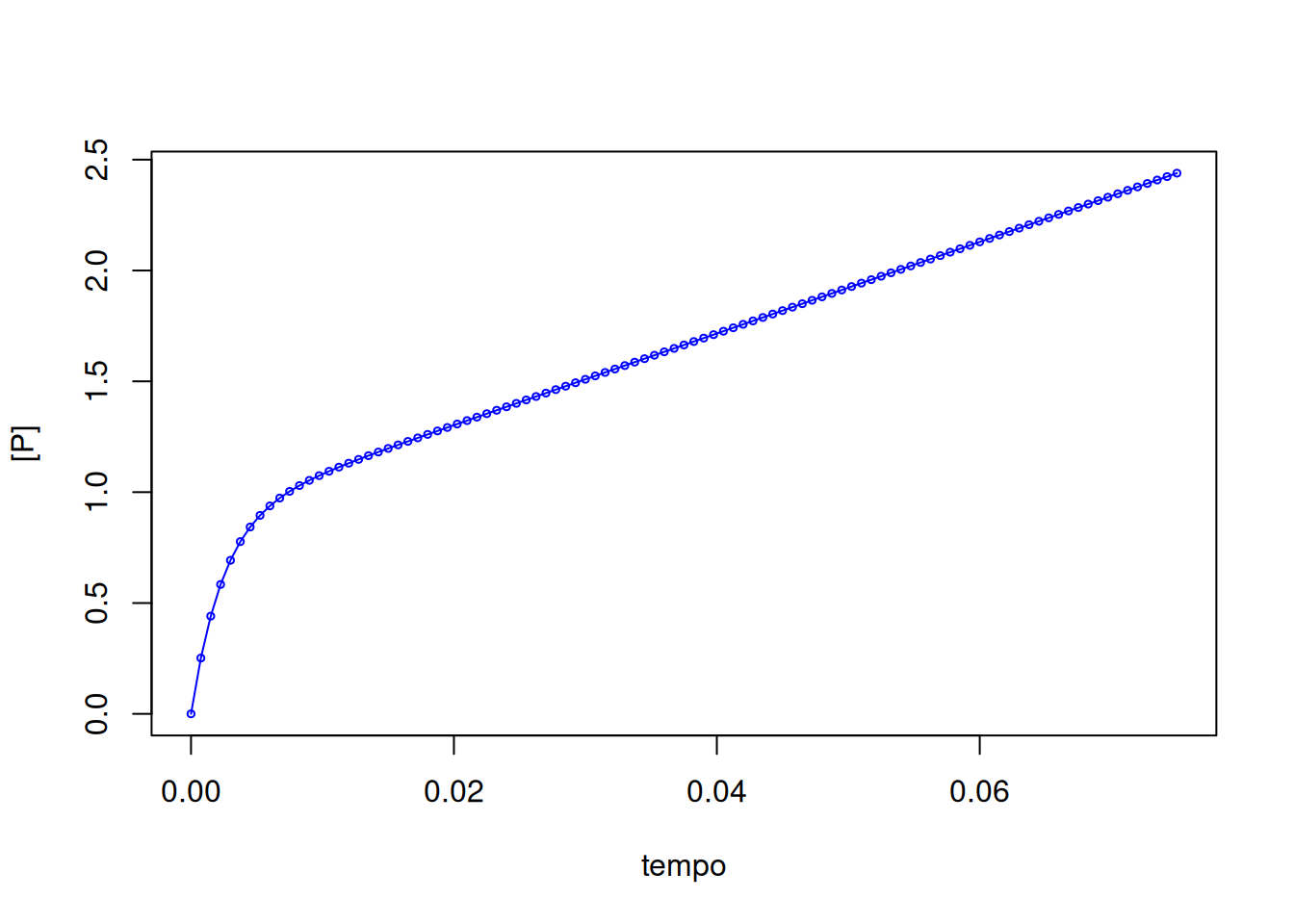
# Curva de MM em enzima de comportamento pré-estacionário
# Parâmetros
k2 <- 387
km2 <- 3
k3 <- 22
xmin <- 0
xmax <- 0.075 # definição de limites para função
# Variáveis da equação de simulação (função dos parâmetros)
kobs <- k2 + km2 + k3
Ao <- k2 * (k2 + km2) / kobs^2
kcat <- k2 * k3 / kobs
# Definição da função de simulação
sim <- function(x, kobs, Ao, kcat) {
Ao * (1 - exp(-kobs * x)) + kcat * x
}
# Curval de simulação
curve(sim(x, kobs = kobs, Ao = Ao, kcat = kcat),
col = "blue",
type = "o", xlim = c(xmin, xmax), cex = 0.5,
xlab = "tempo", ylab = "[P]"
)
R possui funções de minimização que permitem encontrar a raíz de equações lineares ou não lineares.optim em stats ou o pacote rootSolve, que buscam minimizar equações lineares e não lineares para encontrar os valores de seus parâmetros.R com rootSolve, adicionando ainda a busca para Km como segue.\[ Km = \frac{k3}{k2+k3} \] {#eq:burstKm}
# Cálculo de constantes cinéticas por solução de sistema de equações
# não lineares aplicadas à cinética de burst.
library(rootSolve)
kobs <- 0.06
Ao <- 50
kcat <- 300
Ks <- 15
# define os parâmetros de ajuste não linear obtidos por curva progressiva
# experimental, t x P;
# Obs: Ks obtido experimentalmente de curva de S x kobs
# Parâmetros
# x[1]=k2
# x[2]= k3
# x[3] = Km
# Modelo
model <- function(x) c(x[1] / kobs^2 - Ao, (x[1] * x[2]) / kobs - kcat,
Ks * x[2] / (x[1] + x[2]) - x[3])
# o modelo acima deve conter uma lista de equações cuja igualdade é zero,
# ou seja, f(x)=0
(ss <- multiroot(model, c(1, 1, 1))) # comando de execução do rootSolve$root
[1] 0.18000 100.00000 14.97305
$f.root
[1] 0.000000e+00 1.136868e-13 -1.243054e-09
$iter
[1] 4
$estim.precis
[1] 4.143891e-10R na solução de problemas quantitativos em biofísico-química. Dessa forma, omitimos diversos conceitos, tais como cinética lenta de interação de substrato (slow binding), cinética de múltiplos substratos (reação sequencial e ping-pong), equação integrada de Michaelis-Menten e curvas progressivas, ativação de moduladores, influência de pH e temperatura na catálise, e enzimas multisítios, entre vários.